Team:Wash U/Spanish/Biological Parts
From 2009.igem.org
(Difference between revisions)
| (4 intermediate revisions not shown) | |||
| Line 119: | Line 119: | ||
|} | |} | ||
<div align="left"><font size="2"> | <div align="left"><font size="2"> | ||
| - | [[Team:Wash_U/Biological_Parts# | + | [[Team:Wash_U/Spanish/Biological_Parts#Partes|Volver Arriba]] |
<font size="4"> | <font size="4"> | ||
| Line 131: | Line 131: | ||
[[Image:Slide5.jpg|480px]] | [[Image:Slide5.jpg|480px]] | ||
<font size="2"> | <font size="2"> | ||
| - | [[Team:Wash_U/Biological_Parts# | + | [[Team:Wash_U/Spanish/Biological_Parts#Partes|Volver Arriba]] |
<font size="4"> | <font size="4"> | ||
| Line 141: | Line 141: | ||
[[Image:pucBAModelRxn.jpg|thumb|200px|pucBA Model Reactions]] | [[Image:pucBAModelRxn.jpg|thumb|200px|pucBA Model Reactions]] | ||
[[Image:pucBAModelTestSim.jpg|thumb|200px|Test Simulation Output]] | [[Image:pucBAModelTestSim.jpg|thumb|200px|Test Simulation Output]] | ||
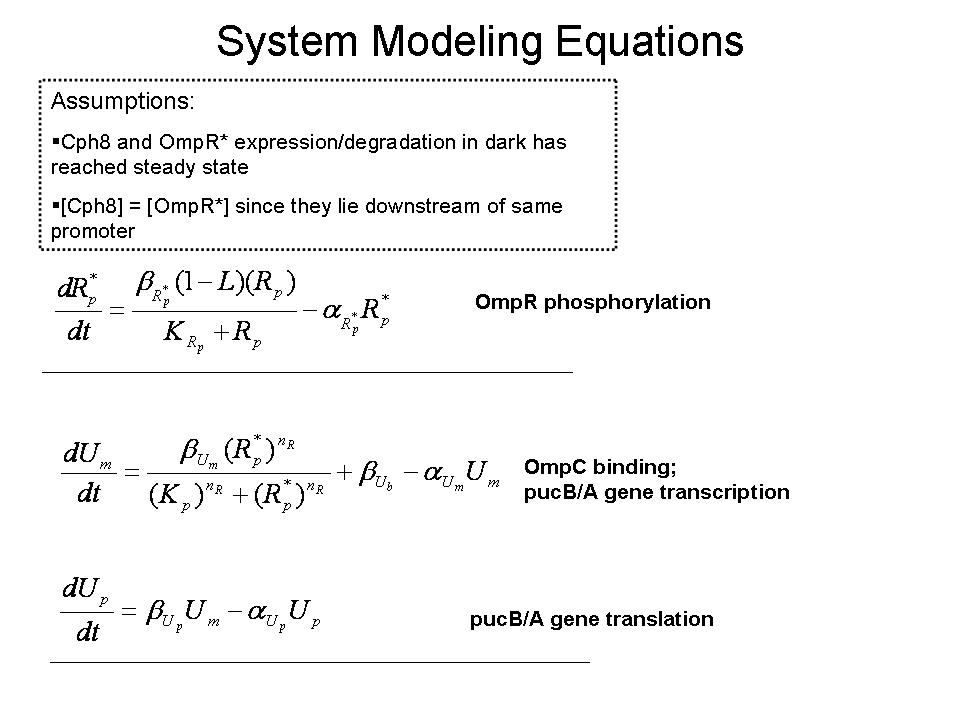
| - | ''' | + | '''Modelado de la red de regulación genética''' |
| - | : | + | :Nuestro grupo pretende evaluar la optimización de la síntesis del sistema que modula pucB / A la expresión de los genes y complejos LH2 montaje''en Rhodobacter sphaeroides''. Aquí se emplean un modelo matemático de este sistema para generar las predicciones sobre el comportamiento de los activos del sistema en respuesta a la luz de entrada. Características del sistema que el modelo puede ayudar a investigar incluyen la escala de tiempo de respuesta a las señales de luz, la solidez del sistema en respuesta a las fluctuaciones en la intensidad de la luz, y la traducción entre los cambios en la expresión génica y la absorbancia del espectro de la ingeniería de células. |
<br> | <br> | ||
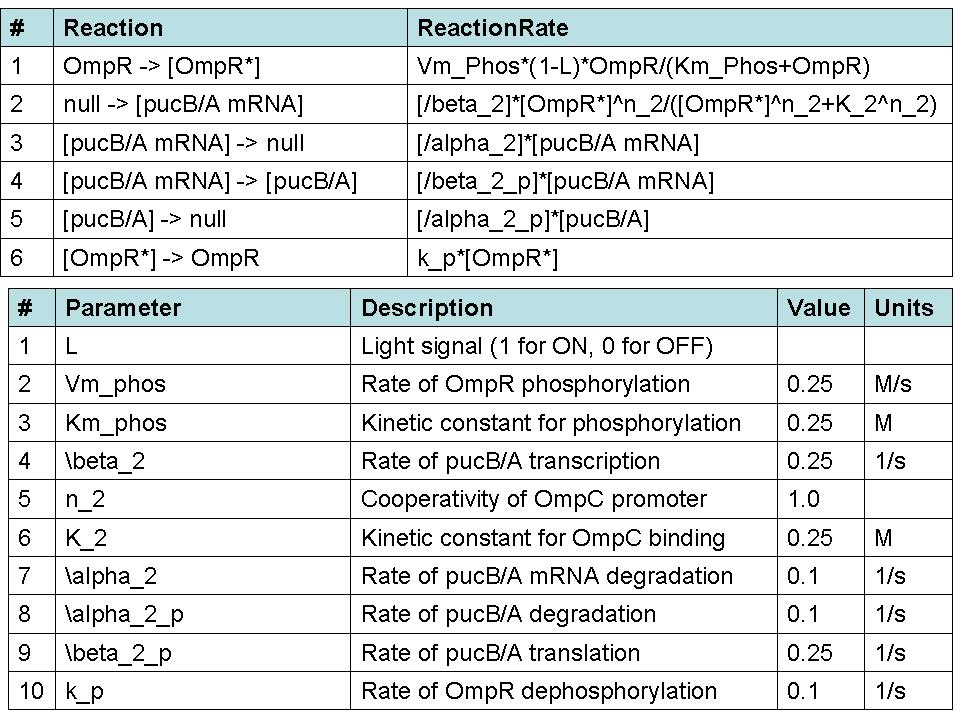
| - | : | + | :Aunque el contexto del modelo puede extenderse a la transcripción de EPRR / B genes implicados en la integración de oxígeno y señales luminosas, un primer modelo de prueba fue desarrollado utilizando las hipótesis de determinadas condiciones iniciales para aislar el efecto de la señal luminosa. Desde Cph8 y OmpR se encuentra en la misma transcripción abajo de la PUC región promotora, se suponía que sus ARNm y proteínas ya había alcanzado el estado estacionario las concentraciones. Además, las concentraciones de los factores que se supone que son iguales en este estado. El modelo cuyo diagrama se construyó en la caja de herramientas distribuidas por Simbiology MathWorks detalles clave de reacciones que conduzcan a la traducción de la pucB / A genes. La ecuación de velocidad de reacción utilizadas para la fosforilación de OmpR como consecuencia de la señal luminosa Cph8 llegar OmpR está obligado a reflejar en una forma modificada de la cinética de Michaelis-Menten. Una lógica de la función que corresponde a la luz ON / OFF (1 / 0) multiplica la máxima velocidad de reacción en el numerador. Así, el modelo asume que no se produce por la fosforilación de este mecanismo en ausencia de luz. El promotor OmpC vinculante ecuación se basa en la ecuación de Hill represor (1). |
<br> | <br> | ||
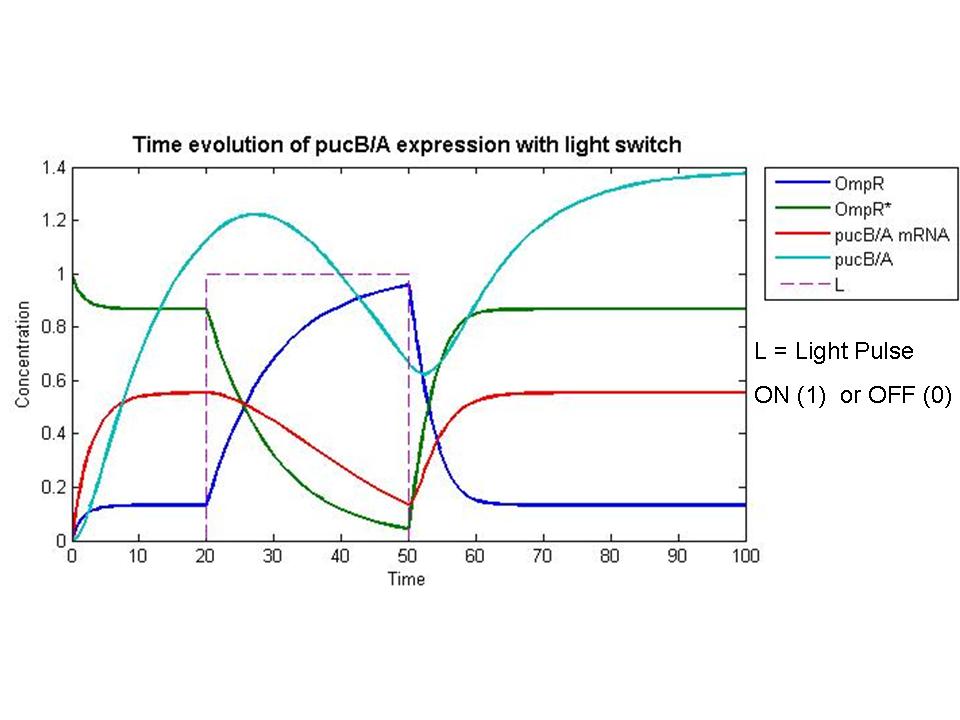
| - | : | + | :Caracterización de componentes y los pasos se están realizando búsquedas en la literatura con el fin de obtener parámetros cuantitativos para la reacción de las tasas. Con el fin de simular el comportamiento del sistema, se incluyeron los valores putativo exagerar cierto que las concentraciones y escalas de tiempo. OmpR se dio una concentración inicial normalizado a uno, y todos los demás componentes fueron asumidos inicialmente insignificante a este valor. Un ideal de pulso de luz se presentó en un instante y se eliminan de simulación treinta segundos después. A partir de este rudimentario de simulación que se puede extraer que el nonlinearities de la fosforilación y factor de transcripción vinculante cinética efectivamente facilitar la entrada de luz fuerte. Por diseño, el interruptor de luz sobre el rendimiento de la fosforilación de OmpR y la represión de la pucB / A los genes que dan lugar a LH2. Por el contrario, cuando dejó, la concentración de pucB / A hasta que se recupere y se aumenta el estado de equilibrio determinado por su traducción y la degradación de las tasas. |
| - | === | + | |
| + | === Referencias === | ||
1. Alon, Uri. <u>Introduction to systems biology and the design principles of biological networks</u>. Boca Raton, FL: Chapman & Hall, 2006. | 1. Alon, Uri. <u>Introduction to systems biology and the design principles of biological networks</u>. Boca Raton, FL: Chapman & Hall, 2006. | ||
| Line 157: | Line 158: | ||
3. <u>System modeling in cellular biology from concepts to nuts and bolts.</u> Cambridge, MA: MIT P, 2006. | 3. <u>System modeling in cellular biology from concepts to nuts and bolts.</u> Cambridge, MA: MIT P, 2006. | ||
| - | <br>[[Team:Wash_U/Biological_Parts# | + | <br>[[Team:Wash_U/Spanish/Biological_Parts#Partes|Volver Arriba]] |
<br> | <br> | ||
{{WashUbottom}} | {{WashUbottom}} | ||
Latest revision as of 19:48, 7 July 2009
Partes
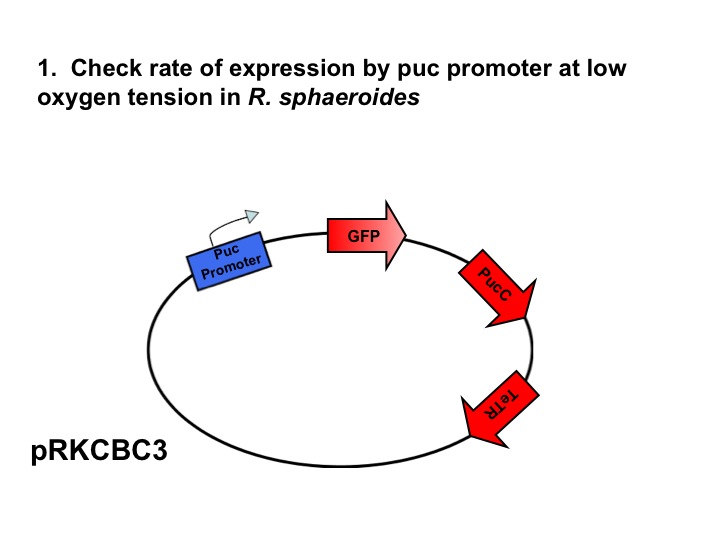
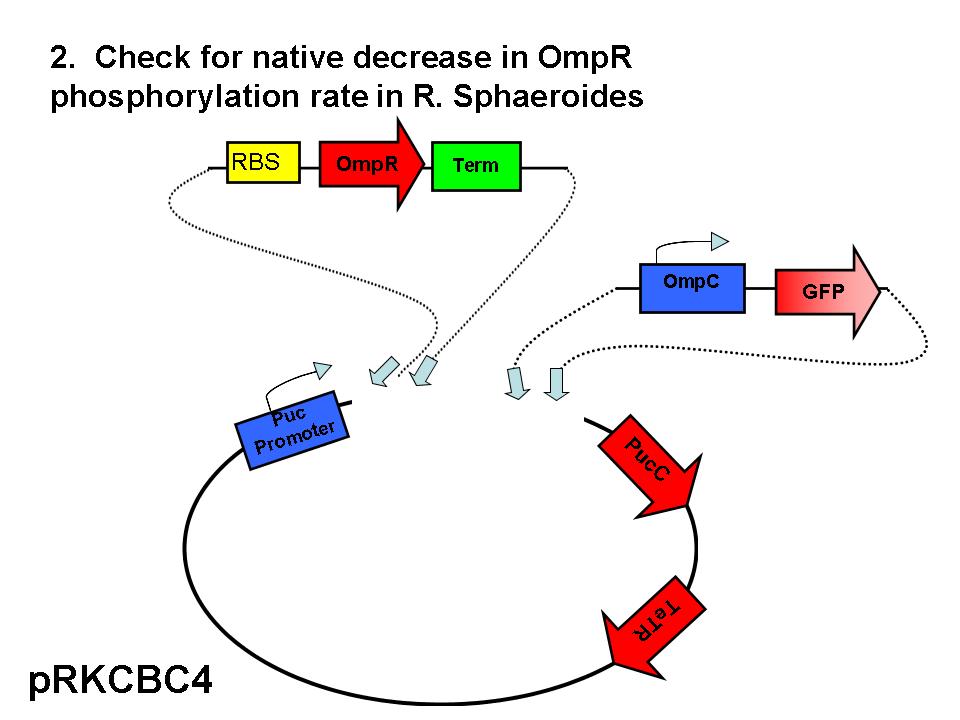
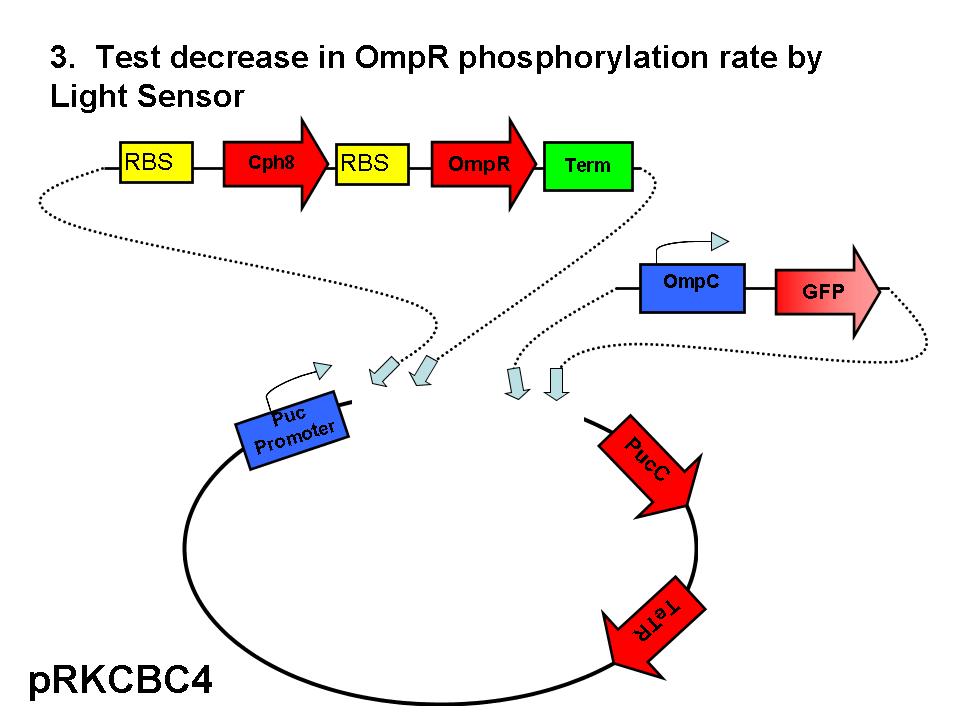
| Component | Description | Part/Accession # | Base Pairs | Plasmid | Resistance | Well |
| RBS | Ribosomal Binding Site | [http://partsregistry.org/Part:BBa_B0034 BBa_B0034] | 12 | pSB1A2 | Ampicillin | plate 1, 2M |
| Red Light Sensor | description | [http://partsregistry.org/Part:BBa_I15010 BBa_I15010] | 2,238 | pSB2K3 | Kanamycin | N/A |
| OmpR (E. coli) | description | [http://www.ncbi.nlm.nih.gov/sites/entrez?Db=gene&Cmd=retrieve&dopt=full_report&list_uids=947913&log$=databasead&logdbfrom=protein NP_417864.1] | 720 | pSB1T3 | Tetracycline | N/A |
| Terminator | Stops Transcription | [http://partsregistry.org/Part:BBa_B0015 BBa_B0015] | 129 | pSB1AK3 | Ampicillin and Kanamycin | plate 1, 23L |
| OmpR + Terminator | description | sequence | 916 | pany-amp | Ampicillin | synthesized |
| OmpC promoter | description | [http://partsregistry.org/Part:BBa_R0082 BBa_R0082] | 108 | pSB1A2 | Ampicillin | plate 1, 16K |
| puc B/A | description | sequence | 375 | pSB1A3 | Ampicillin | N/A |
| puc B | description | [http://www.ncbi.nlm.nih.gov/sites/entrez?Db=gene&Cmd=retrieve&dopt=full_report&list_uids=3719170&log$=databasead&logdbfrom=nuccore YP_353390] | 156 | ? | ? | N/A |
| puc A | description | [http://www.ncbi.nlm.nih.gov/sites/entrez?Db=gene&Cmd=retrieve&dopt=full_report&list_uids=3719171&log$=databasead&logdbfrom=nuccore YP_353391] | 165 | ? | ? | N/A |
| OmpC promoter+BA | description | sequence | 539 | pany-kana | Kanamycin | synthesized |
| Light Response System | description | [http://partsregistry.org/Part:BBa_M30109 BBa_M30109] | 4,333 | ? | Ampicillin | N/A |
| TetR repressible | description | [http://partsregistry.org/Part:BBa_J13002 BBa_J13002] | 74 | pSB1A2 | Ampicillin | plate 1, 13B |
| Green Flourescent Protein | Marker for successful transformation | [http://partsregistry.org/Part:BBa_E0240 BBa_E0240] | 976 | pSB1A2 | Ampicillin | plate 1, 12M |
Caracterizacion
Modelado
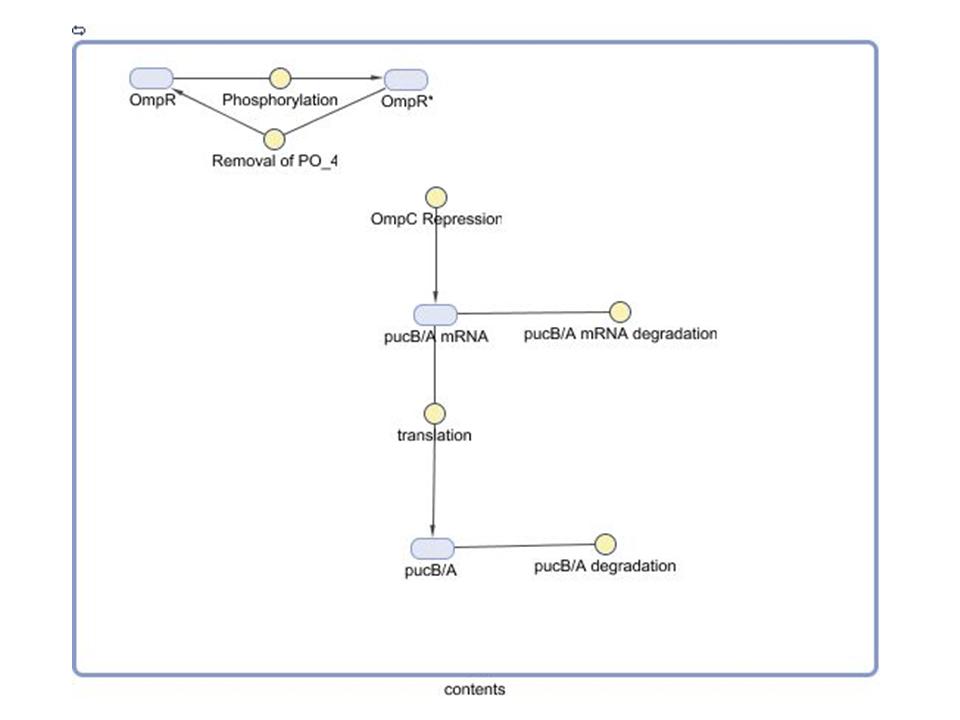
Modelado de la red de regulación genética
- Nuestro grupo pretende evaluar la optimización de la síntesis del sistema que modula pucB / A la expresión de los genes y complejos LH2 montajeen Rhodobacter sphaeroides. Aquí se emplean un modelo matemático de este sistema para generar las predicciones sobre el comportamiento de los activos del sistema en respuesta a la luz de entrada. Características del sistema que el modelo puede ayudar a investigar incluyen la escala de tiempo de respuesta a las señales de luz, la solidez del sistema en respuesta a las fluctuaciones en la intensidad de la luz, y la traducción entre los cambios en la expresión génica y la absorbancia del espectro de la ingeniería de células.
- Aunque el contexto del modelo puede extenderse a la transcripción de EPRR / B genes implicados en la integración de oxígeno y señales luminosas, un primer modelo de prueba fue desarrollado utilizando las hipótesis de determinadas condiciones iniciales para aislar el efecto de la señal luminosa. Desde Cph8 y OmpR se encuentra en la misma transcripción abajo de la PUC región promotora, se suponía que sus ARNm y proteínas ya había alcanzado el estado estacionario las concentraciones. Además, las concentraciones de los factores que se supone que son iguales en este estado. El modelo cuyo diagrama se construyó en la caja de herramientas distribuidas por Simbiology MathWorks detalles clave de reacciones que conduzcan a la traducción de la pucB / A genes. La ecuación de velocidad de reacción utilizadas para la fosforilación de OmpR como consecuencia de la señal luminosa Cph8 llegar OmpR está obligado a reflejar en una forma modificada de la cinética de Michaelis-Menten. Una lógica de la función que corresponde a la luz ON / OFF (1 / 0) multiplica la máxima velocidad de reacción en el numerador. Así, el modelo asume que no se produce por la fosforilación de este mecanismo en ausencia de luz. El promotor OmpC vinculante ecuación se basa en la ecuación de Hill represor (1).
- Caracterización de componentes y los pasos se están realizando búsquedas en la literatura con el fin de obtener parámetros cuantitativos para la reacción de las tasas. Con el fin de simular el comportamiento del sistema, se incluyeron los valores putativo exagerar cierto que las concentraciones y escalas de tiempo. OmpR se dio una concentración inicial normalizado a uno, y todos los demás componentes fueron asumidos inicialmente insignificante a este valor. Un ideal de pulso de luz se presentó en un instante y se eliminan de simulación treinta segundos después. A partir de este rudimentario de simulación que se puede extraer que el nonlinearities de la fosforilación y factor de transcripción vinculante cinética efectivamente facilitar la entrada de luz fuerte. Por diseño, el interruptor de luz sobre el rendimiento de la fosforilación de OmpR y la represión de la pucB / A los genes que dan lugar a LH2. Por el contrario, cuando dejó, la concentración de pucB / A hasta que se recupere y se aumenta el estado de equilibrio determinado por su traducción y la degradación de las tasas.
Referencias
1. Alon, Uri. Introduction to systems biology and the design principles of biological networks. Boca Raton, FL: Chapman & Hall, 2006.
2. Bower, James M. Computational Modeling of Genetic and Biochemical Networks (Computational Molecular Biology). New York: M.I.T. PRESS, 2001.
3. System modeling in cellular biology from concepts to nuts and bolts. Cambridge, MA: MIT P, 2006.

|
 "
"