Team:Chiba/Project
From 2009.igem.org
(Difference between revisions)
(→Conclusions) |
(→Conclusions) |
||
| Line 133: | Line 133: | ||
== Conclusions == | == Conclusions == | ||
| - | + | *By using error-prone PCR, we have created a LuxR library. | |
| - | + | *With a simple and convenient screening method, we have isolated various LuxR mutants which confer delayed switching behavior in GFP signals. | |
| - | + | *By introducing these LuxR variants together with reporter genes (such as GFP) under the control of Lux promoter, we created bacteria 'ink's that develop their color with unique delay-time. | |
| - | + | *We conducted painting with these bacteria inks thereby created animated pictures. | |
| - | *error- | + | *Characterization of LuxR mutants is ongoing. But our preliminary data showed that some of the variants turned out to be the one with less sensitivity to AHLs, and others seemed to be as sensitive as wild-type LuxR but seemed less efficient somewhere in the downstream process. |
| - | + | *We created variety of Biobricks during the course of this projects. Some of them are characterized and sent to the HQ, and many more are almost ready for shipping! | |
| - | * | + | |
| - | * | + | |
| - | + | ||
| - | + | ||
| - | + | ||
| - | * | + | |
| - | + | ||
| - | * | + | |
| - | + | ||
| - | + | ||
| - | + | ||
|} | |} | ||
|} | |} | ||
Revision as of 08:15, 21 October 2009
| Home | The Team | Parts | Reference | Notebook | Protocols | Links | Acknowledgements | Contact |
|---|
|
IntroductionMaking a "bacterial timer"!
(手術予定)
そして、昨年は2段階の通信しか創れなかった。バリエーションが乏しい。
Project Design(手術予定) 2) Project Design ⑤Receiver側のAHL通信の仕組みを図説する。(1,AHLが入るところ(培地調節) 2,Receiver中のLuxR 3,Receiver中のReporter)
Experiments, Results & DiscussionLuxR mutantづくり
Experiments
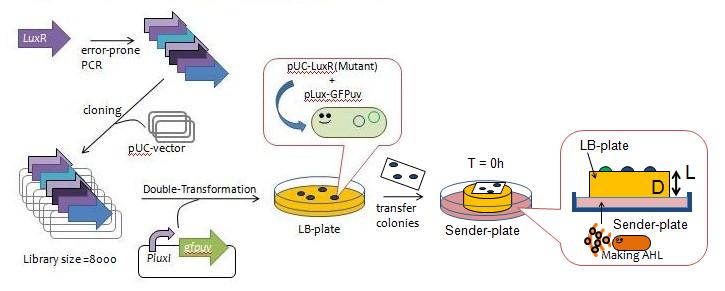
(手術予定) ⑥Mutantを創る。(Error-proneが良い理由:バリエーションを創りやすい。新たなBiobrick作成方法としてError-prone PCRとスクリーニングをセットにしMutant-Partsの作り方を説明する。) ⑦さらに詳しい実験方法(Biobrickからerror-prone PCRをおこなったことなど) ⑧得られたデーター(なぜ8000のライブラリを、200に絞り、どうやって13sampleを選び出したのか説明) 得られたLuxR mutantのcharacterization⑨LuxR Mutant 個性確定実験の実験方法
⑩100 nM培地での応答の遅れについての結果
⑫LuxR Mutantの個性と、変位が入っている部分からの考察
Demonstration⑯E. coli Timer完成デモ 明日のよるにはあげられます。--Yoshimi 14:21, 20 October 2009 (UTC) (⑰できればアニメ) For the determination of best condition for E coli painting, Click here! Conclusions
|
|}
 "
"