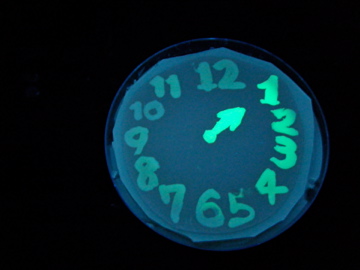
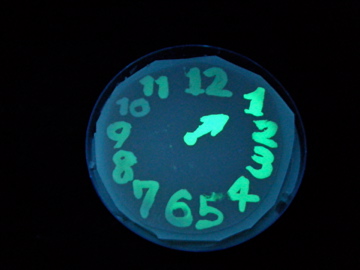
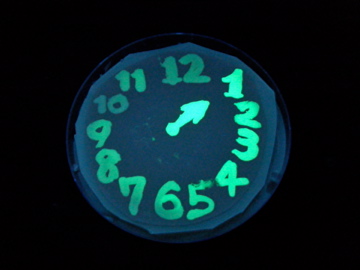
|
|
Introduction
Implementing a "Timer" Function!
- Our project is to make a sort of "timer", where gene activation is triggered after a certain time.
- Our approach to this goal is to create a series of transcription factors (TFs) that can be activated by the same inducer molecule but with a different sensitivity. In an environment where the inducer concentration gradually levels up, these TFs switches on one by one according to the order of sensitivity.
- We believe such collection of such TF variants would be useful for the timing control in biological function at will, and thereby contribute to the synthetic biology & iGEM community.
- Using a set of TF variants, we aimed to draw an "animated picture": a picture that pop up one by one.
|
|
Project Design
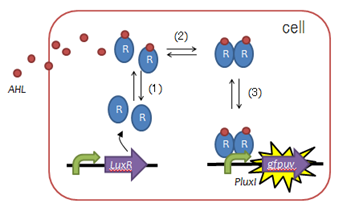 Fig. Function of LuxR :(1)LuxR protein generation, (2)AHL binding domain, (3)dimerization, (4)DNA binding domain - 私たちはタイマーを創るためにLuxRを利用する。
- LuxRはcell-cellcommunicationで使われるタンパクである。
- LuxRは細胞内で左図にしめすとおりに働く。
- 私たちはLuxRに変異を入れることで、スイッチングのdelayを数種類入れられると考えた。
Experiments, Results & Discussion
Experiments
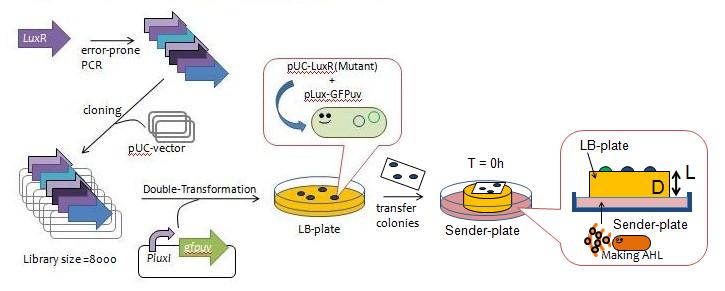 Fig. Y Directed evolution to get some delayed-LuxR mutants - error-prone PCRでLuxRの変異ライブラリ(ライブラリサイズ=8000)を作製し,発現ベクターに組み込んだ
- これを,E.coli BW1226 harboring plux-gfpに形質転換し,200のコロニーを形成させた。
- コロニーをニトロセルロース膜でリフトし,AHL入りのプレートにのせ,gfpの蛍光の経時変化をみた(Fig. X)。
- 蛍光しはじめるのが遅いものを13個pickし,delayed-LuxR変異体を得た。
 Fig. X LuxRライブラリによるのPlux-gfp蛍光経時変化
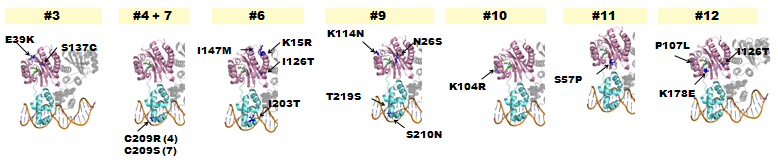 Fig. - リガンド(AHL)結合ドメイン/DNA結合ドメイン、どちらに変異が入っても、Delay変異体の表現型を与えた。
Characterization of LuxR Mutants
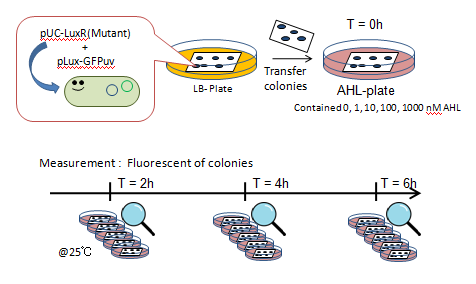 Fig. Characterization of LuxR Mutants Experiments
- LuxR WT, Mutant(13種)とpLux-GFPのプラスミドが2つトランスフォーメーションされたJW1226株を12h@37℃液体培養。
- Cultureを48ピンでNCフィルタへ植菌し、フィルタを固体培地にのせて12h@37℃で培養。
- AHLの濃度を0, 1, 10, 100, 1000 nMでふった固体培地に、NCフィルタを移動。
- この時を測定開始点とし、30分毎に4時間蛍光の上がり方を目視で確認。
- フナコシのUVGL-58のlong waveは365 nm
Results and Discussion
100 nM培地での応答の遅れについての結果
 Fig.時間依存 - 一番GFP強度の上がり方がばらついた100 nM AHL培地の写真。
- 応答速度の異なる以下のようなタイプのMutantを確認できた。
- スイッチが入るタイミングが違うモノ
- スイッチが入ってもなかなか蛍光強度が上がらないモノ
AHL濃度を振った場合の、6h後の蛍光強度の差についての結果
 Fig.濃度依存 - スイッチングが起こる濃度がWTと異なるMutantを得ることができた。
- 変異がはいっている部分がほとんど同じである#4と#7の間に、以下のような差が見られた。
- 4 : 10 nM AHLで蛍光がみられる。
- 7 : 100 nM AHLで蛍光がみられる。
Demonstration
- 得られたMutantを使ってデモンストレーション実験を行った。
- For the determination of best condition for E coli painting, Click here!
Conclusions
- By using error-prone PCR, we have created a LuxR library.
- With a simple and convenient screening method, we have isolated various LuxR mutants which confer delayed switching behavior in GFP signals.
- By introducing these LuxR variants together with reporter genes (such as GFP) under the control of Lux promoter, we created bacteria 'ink's that develop their color with unique delay-time.
- We conducted painting with these bacteria inks thereby created animated pictures.
- Characterization of LuxR mutants is ongoing. But our preliminary data showed that some of the variants turned out to be the one with less sensitivity to AHLs, and others seemed to be as sensitive as wild-type LuxR but seemed less efficient somewhere in the downstream process.
- We created variety of Biobricks during the course of this projects. Some of them are characterized and sent to the HQ, and many more are almost ready for shipping!
|
 "
"