Team:SupBiotech-Paris/Ciblage Cellulaire
From 2009.igem.org
(→Résultats [1,2]) |
|||
| (18 intermediate revisions not shown) | |||
| Line 6: | Line 6: | ||
== Contexte == | == Contexte == | ||
| - | Après l’action du [[Team:SupBiotech-Paris/Concept1Fr#drapeau|vecteur tissulaire]], viens | + | Après l’action du [[Team:SupBiotech-Paris/Concept1Fr#drapeau|vecteur tissulaire]], viens celle du [[Team:SupBiotech-Paris/Concept2Fr#drapeau|vecteur cellulaire]]. Ce dernier est un bactériophage modifié qui a la faculté d’infecter les cellules eucaryotes. Le bactériophage lambda, du fait de sa grande capacité de clonage et une structure de capside adaptée à une présence concentrée de protéines exogènes, est un très bon candidat pour le design d’un [[Team:SupBiotech-Paris/Concept1Fr#drapeau|vecteur tissulaire]] eucaryote. La [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]] issue de la capside de l’adénovirus apparait comme un candidat prometteur pour le ciblage du phage lambda. En effet, elle est dotée de plusieurs fonctions telles que la liaison aux récepteurs cellulaires, l’internalisation des particules virales et la libération de la capside par l’endosome.<br> |
| + | |||
| + | |||
| + | <span style="float: right">[[Team:SupBiotech-Paris/Ciblage_Cellulaire#drapeau|Haut de page]]</span> | ||
==Objectif == | ==Objectif == | ||
| - | Nos objectifs sont de designer un [[Team:SupBiotech-Paris/Concept2Fr#drapeau|vecteur cellulaire]] de type bactériophage Lambda recombiné avec une base de penton issue de l’adénovirus 5 fusionnée à sa [[Team:SupBiotech-Paris/BiobricksFr#drapeau|protéine D]]. Le [[Team:SupBiotech-Paris/Concept1Fr#drapeau|vecteur tissulaire]] doit être capable d’intégrer la cellule, sortir de l’endosome, transporter son ADN vers le noyau de la cellule et finalement transcrire ce(s) [[Team:SupBiotech-Paris/Concept3Fr#drapeau|gène(s) thérapeutique(s)]]. <br> | + | Nos objectifs sont de designer un [[Team:SupBiotech-Paris/Concept2Fr#drapeau|vecteur cellulaire]] de type bactériophage Lambda recombiné avec une [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]] issue de l’adénovirus 5 fusionnée à sa [[Team:SupBiotech-Paris/BiobricksFr#drapeau|protéine D]]. Le [[Team:SupBiotech-Paris/Concept1Fr#drapeau|vecteur tissulaire]] doit être capable d’intégrer la cellule, sortir de l’endosome, transporter son ADN vers le noyau de la cellule et finalement transcrire ce(s) [[Team:SupBiotech-Paris/Concept3Fr#drapeau|gène(s) thérapeutique(s)]]. <br> |
| Line 17: | Line 20: | ||
== Démarche expérimentale == | == Démarche expérimentale == | ||
| - | Dans le cadre du design des gènes du bactériophage recombinant nous avons décidé de fusionner la base de penton de l’adénovirus 5 avec la [[Team:SupBiotech-Paris/BiobricksFr#drapeau|protéine D]] du phage lambda. L’extraction de la protéine D à partir du génome du bactériophage Lambda a été menée par réaction de polymérisation en chaine (PCR) avec plusieurs paires de primers. La même stratégie a été prise pour l’extraction de la base de penton de l’adénovirus 5 qui a été extraite d’un plasmide codant pour le virus gracieusement donné par le Dr. Karim Benihoud (UMR8121, CNRS/IGR, Villejuif, France). <br> | + | Dans le cadre du design des gènes du bactériophage recombinant nous avons décidé de fusionner la [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]] de l’adénovirus 5 avec la [[Team:SupBiotech-Paris/BiobricksFr#drapeau|protéine D]] du phage lambda. L’extraction de la protéine D à partir du génome du bactériophage Lambda a été menée par réaction de polymérisation en chaine (PCR) avec plusieurs paires de primers. La même stratégie a été prise pour l’extraction de la [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]] de l’adénovirus 5 qui a été extraite d’un plasmide codant pour le virus gracieusement donné par le Dr. Karim Benihoud (UMR8121, CNRS/IGR, Villejuif, France). <br> |
Après la formation de la protéine de fusion, celle-ci est introduite dans un plasmide BioBrick. Le plasmide contient une résistance contre un antibiotique pour la confirmation de la transfection du phage recombiné dans la bactérie. Ainsi qu’un gène rapporteur tel que la GFP avec un promoteur eucaryote, le CMV du <i>Simian virus</i> 40 (SV40), pour confirmer la transfection dans les cellules eucaryotes. Cette stratégie nous permet alors de prouver que le bactériophage est capable d’infecter les cellules eucaryotes. <br> | Après la formation de la protéine de fusion, celle-ci est introduite dans un plasmide BioBrick. Le plasmide contient une résistance contre un antibiotique pour la confirmation de la transfection du phage recombiné dans la bactérie. Ainsi qu’un gène rapporteur tel que la GFP avec un promoteur eucaryote, le CMV du <i>Simian virus</i> 40 (SV40), pour confirmer la transfection dans les cellules eucaryotes. Cette stratégie nous permet alors de prouver que le bactériophage est capable d’infecter les cellules eucaryotes. <br> | ||
| - | Malheureusement nous n’avons pas été capable de construire la protéine de fusion dans le temps requis. Cependant la littérature scientifique démontre que la confection d’un [[Team:SupBiotech-Paris/Concept1Fr#drapeau|vecteur tissulaire]] type bactériophage lambda est possible par fusion de la base de penton de l’adénovirus avec la [[Team:SupBiotech-Paris/BiobricksFr#drapeau|protéine D]] (Stefania Piersanti et al. 2004). La séquence centrale de la | + | Malheureusement nous n’avons pas été capable de construire la protéine de fusion dans le temps requis. Cependant la littérature scientifique démontre que la confection d’un [[Team:SupBiotech-Paris/Concept1Fr#drapeau|vecteur tissulaire]] type bactériophage lambda est possible par fusion de la [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]] de l’adénovirus avec la [[Team:SupBiotech-Paris/BiobricksFr#drapeau|protéine D]] (Stefania Piersanti et al. 2004). La séquence centrale de la [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]], acides aminés 1 à 571, fusionnée avec le bactériophage offre une transfection dans les cellules eucaryotes, tous comme l’utilisation du fragment RGD responsable de l’entrée du virus et la sortie de l’endosome, fragment 286 à 393. <br> |
| Line 30: | Line 33: | ||
=== Design de la protéine de fusion === | === Design de la protéine de fusion === | ||
| - | Pour le design de la protéine de fusion, nous avons décidé d’extraire séparément la base de penton de l’adénovirus 5 | + | Pour le design de la protéine de fusion, nous avons décidé d’extraire séparément la [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]] de l’adénovirus 5 et la [[Team:SupBiotech-Paris/BiobricksFr#drapeau|protéine D]] du bactériophage lambda grâce à des primers qui contiennent un site de restriction BalI sur le primer reverse de la [[Team:SupBiotech-Paris/BiobricksFr#drapeau|protéine D]] et le primer forward de la [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]]. De plus la protéine de fusion finale contient les fragments spécifiques aux BioBricks à ces deux extrémités. <br> |
Pour l’extraction des 2 gènes nous avons utilisé les primers suivants : <br> | Pour l’extraction des 2 gènes nous avons utilisé les primers suivants : <br> | ||
| Line 39: | Line 42: | ||
Protéine D du phage Lambda: <br> | Protéine D du phage Lambda: <br> | ||
| - | Forward : ATG-ACG-AGC-AAA-GAA-ACC-TT; <br> | + | Forward : 5' ATG-ACG-AGC-AAA-GAA-ACC-TT 3'; <br> |
| - | Reverse : AAA-AAA-ATC-CCG-TAA-AAA-AAG-C. <br> | + | Reverse : 5' AAA-AAA-ATC-CCG-TAA-AAA-AAG-C 3'. <br> |
Base de penton de l’adénovirus 5 : <br> | Base de penton de l’adénovirus 5 : <br> | ||
| - | Forward : AAT-GGC-CAA-TGC-GGC-GCG-CGG-CGA-TG <br> | + | Forward : 5' AAT-GGC-CAA-TGC-GGC-GCG-CGG-CGA-TG 3' <br> |
| - | Reverse : CTG-CAG-CGG-CCG-CTA-CTA-GTA-TCA-AAA-AGT-GCG-G <br> | + | Reverse : 5' CTG-CAG-CGG-CCG-CTA-CTA-GTA-TCA-AAA-AGT-GCG-G 3' <br> |
| Line 51: | Line 54: | ||
| - | Forward : CGA-AAA-AAA-TGC-CCT-AAA-AAA-AAC-CGG-T <br> | + | Forward : 5' CGA-AAA-AAA-TGC-CCT-AAA-AAA-AAC-CGG-T 3' <br> |
| - | Reverse : AAT-GGC-CAA-AAA-AAA-TCC-CGT-AAA-AAA-AGC <br> | + | Reverse : 5' AAT-GGC-CAA-AAA-AAA-TCC-CGT-AAA-AAA-AGC 3' <br> |
| Line 58: | Line 61: | ||
| - | Forward : CTT-AAG-CGC-CGG-CGA-AGA-TC <br> | + | Forward : 5' CTT-AAG-CGC-CGG-CGA-AGA-TC 3' <br> |
| - | Reverse : CTG-CAG-CGG-CCG-CTA-CTA-GTA <br><br> | + | Reverse : 5' CTG-CAG-CGG-CCG-CTA-CTA-GTA 3' <br><br> |
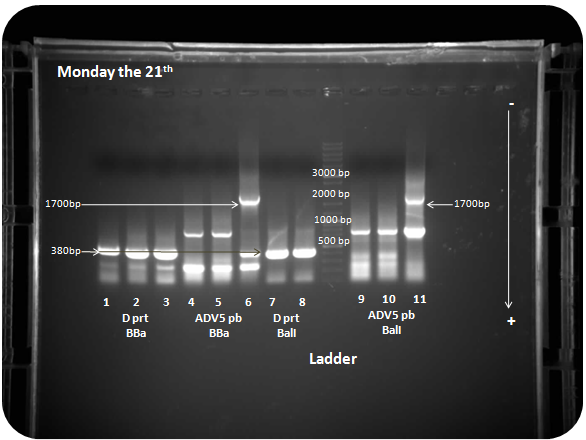
| - | Les résultats de PCR sont présentés dans la figure | + | Les résultats de PCR sont présentés dans la figure 1. Nous observons qu’il y a bien amplification de fragments qui correspondent aux tailles de la [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]] = 1715bp pour l’échantillon 11 et la de protéine D = 385bp pour l’échantillon 7 et 8. Il y a cependant beaucoup de phénomènes de mismatch pendant les cycles d’amplification. Cela pourrait avoir un effet négatif sur le résultat d’amplification final. <br> |
[[image:M2109.png|center]] | [[image:M2109.png|center]] | ||
| - | <i>Figure 1: PCR des BioBricks de la protéine D (1, 2, 3) et de la base de penton (4, 5, 6), de la protéine D ( | + | <i>Figure 1: PCR des BioBricks de la protéine D (1, 2, 3) et de la base de penton (4, 5, 6), de la protéine D (7 et 8) et de la base de penton (9, 10, 11) avec les sites BalI </i><br> |
| Line 72: | Line 75: | ||
=== Transfection des cellules eucaryotes par le phage lambda recombiné avec la base de penton fusionnée à la protéine D (Stefania Piersanti et al., 2004) === | === Transfection des cellules eucaryotes par le phage lambda recombiné avec la base de penton fusionnée à la protéine D (Stefania Piersanti et al., 2004) === | ||
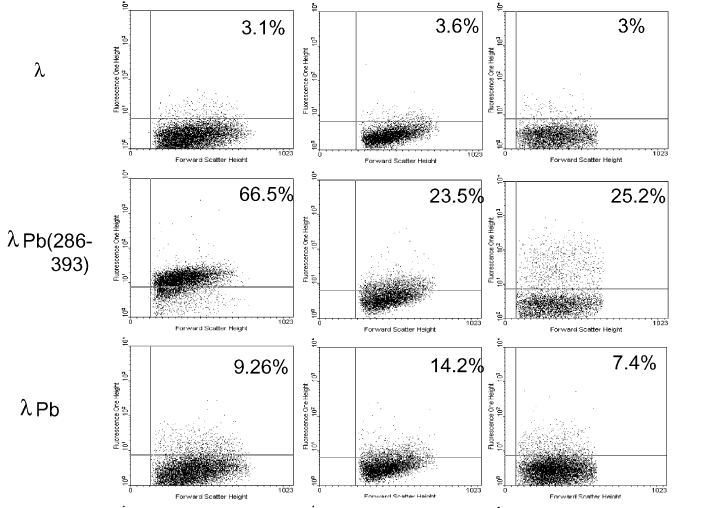
| - | Une étude au par cytofluorimétrie a été faite afin d’analyser le taux de transfection des bactériophages lambda recombinés. La figure | + | Une étude au par cytofluorimétrie a été faite afin d’analyser le taux de transfection des bactériophages lambda recombinés. La figure 2 montre les résultats de cytofluorimétrie de l’analyse de cellules COS-1 après avoir été exposées à une concentration de 10^6 PFU/cellules de phages recombinants, Pb (1-571) ou Pb (286-393). |
[[image:VT1.png|center]] | [[image:VT1.png|center]] | ||
| Line 78: | Line 81: | ||
[[image:VT2.png|center]] | [[image:VT2.png|center]] | ||
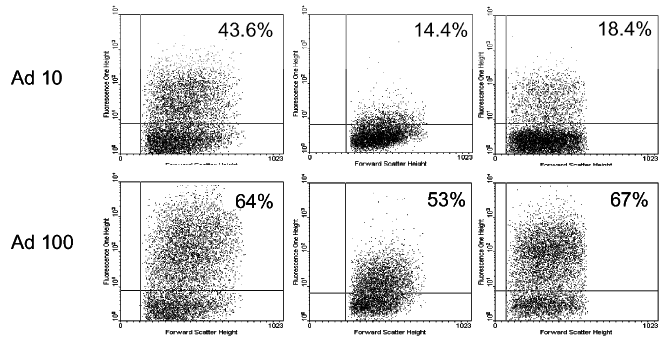
| - | <i> Figure 2 : Analyse de la fluorescence de la GFP sur des phages lambda non recombinés (Lambda), des phages lambda recombinés avec le fragment 286-393 de la base de penton (LambdaPb286-393), des phages lambda recombinés avec la base de penton complète (1-571), des adénovirus marqués à la GFP (Ad10 et Ad100)</i><br> | + | <i> Figure 2 : Analyse de la fluorescence de la GFP sur des phages lambda non recombinés (Lambda), des phages lambda recombinés avec le fragment 286-393 de la [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]] (LambdaPb286-393), des phages lambda recombinés avec la [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]] complète (1-571), des adénovirus marqués à la GFP (Ad10 et Ad100)</i><br> |
| - | Premièrement, nous observons que le phage recombiné montre bien une différence de marquage quelque soit le fragment de base de penton utilisé comparé au bactériophage non transformé. Secondement, le phage recombiné avec le fragment RGD seul (286-393) à une fluorescence plus élevée que le phage avec un fragment complet et plus proche de celui des adénovirus (figure | + | Premièrement, nous observons que le phage recombiné montre bien une différence de marquage quelque soit le fragment de [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]] utilisé comparé au bactériophage non transformé. Secondement, le phage recombiné avec le fragment RGD seul (286-393) à une fluorescence plus élevée que le phage avec un fragment complet et plus proche de celui des adénovirus (figure 3). <br> |
| Line 88: | Line 91: | ||
== Discussion == | == Discussion == | ||
| - | Bien que le vecteur tissulaire n’ait pas été fini, la littérature scientifique montre que la création d’un phage recombiné avec une protéine codant la base de penton de l’adénovirus est possible. Il est aussi démontré que les fragments codant pour les séquences RGD seuls ont une plus forte capacité à infecter les cellules eucaryotes comparé au fragment complet de la base de penton (figure 2). Dans le cas de notre application il est alors possible d’utiliser un bactériophage lambda recombiné pour insérer notre gène thérapeutique. <br> | + | Bien que le vecteur tissulaire n’ait pas été fini, la littérature scientifique montre que la création d’un phage recombiné avec une protéine codant la [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]] de l’adénovirus est possible. Il est aussi démontré que les fragments codant pour les séquences RGD seuls ont une plus forte capacité à infecter les cellules eucaryotes comparé au fragment complet de la [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]] (figure 2). Dans le cas de notre application il est alors possible d’utiliser un bactériophage lambda recombiné pour insérer notre gène thérapeutique. <br> |
| Line 95: | Line 98: | ||
== Conclusions == | == Conclusions == | ||
| - | Pour conclure le fragment RGD seul de la base de penton a la meilleure efficacité d’interaction avec les intégrines des cellules eucaryotes | + | Pour conclure le fragment RGD seul de la [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]] a la meilleure efficacité d’interaction avec les intégrines des cellules eucaryotes. Cependant dans le cadre de notre projet il est plus judicieux d’utiliser la séquence complète de la [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]] (fragment 1-571) car l’utilisation du [[Team:SupBiotech-Paris/Concept2Fr#drapeau|vecteur cellulaire]] et du système d’induction par la doxycycline donne une injection très rapide et très ciblée des bactériophages. L’utilisation d’un système de transfection hautement efficace est déconseillé car les phages n’ont pas le temps de se disperser correctement et vont alors infecter plusieurs fois la même cellule. L’utilisation du fragment complet de la [[Team:SupBiotech-Paris/Concept2Fr#PB|base de penton]] est suffisant pour que le phage infecte correctement les cellules eucaryotes et lui laisse le temps d’avoir une dispersion plus que correct. <br> |
| Line 106: | Line 109: | ||
Dans le cancer du poumon non à petites cellules, ou NSCLC, comme dans tous cancers, la perte de la capacité apoptotique des cellules tumorales est du à la perte fonctionnelle de divers suppresseurs de tumeur entrant dans la voie de signalisation de la cascade apoptotique.<br> | Dans le cancer du poumon non à petites cellules, ou NSCLC, comme dans tous cancers, la perte de la capacité apoptotique des cellules tumorales est du à la perte fonctionnelle de divers suppresseurs de tumeur entrant dans la voie de signalisation de la cascade apoptotique.<br> | ||
| - | L’application du [[Team:SupBiotech-Paris/Introduction1Fr#DVS|DVS]] dans la lutte anti-cancer repose sur le fait de réactiver cette cascade apoptotique en apportant au sein des cellules tumorales une version wild-type des gènes codant | + | L’application du [[Team:SupBiotech-Paris/Introduction1Fr#DVS|DVS]] dans la lutte anti-cancer repose sur le fait de réactiver cette cascade apoptotique en apportant au sein des cellules tumorales une version wild-type des gènes codant la version saine des suppresseurs de tumeur non-fonctionnels.<br> |
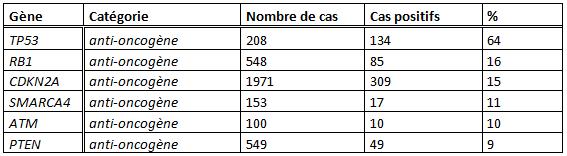
C’est le [http://www.sanger.ac.uk/genetics/CGP/cosmic/ projet COSMIC] de [http://www.sanger.ac.uk/ l’institut Sanger] qui nous a permis de déterminer quels gènes apporter au [[Team:SupBiotech-Paris/Concept3Fr#drapeau|plasmide thérapeutique]] dans le cadre du cancer du poumon à non petites cellules. Ce projet répertorie en effet toutes les mutations détectées pour chaque type de cancers suivant leur fréquence d’apparition. Ainsi, d’après leurs données, la perte de la capacité apoptotique des cellules tumorales pour un cancer du poumon peut être du à la perte fonctionnelle des protéines issus des gènes suivant :<br> | C’est le [http://www.sanger.ac.uk/genetics/CGP/cosmic/ projet COSMIC] de [http://www.sanger.ac.uk/ l’institut Sanger] qui nous a permis de déterminer quels gènes apporter au [[Team:SupBiotech-Paris/Concept3Fr#drapeau|plasmide thérapeutique]] dans le cadre du cancer du poumon à non petites cellules. Ce projet répertorie en effet toutes les mutations détectées pour chaque type de cancers suivant leur fréquence d’apparition. Ainsi, d’après leurs données, la perte de la capacité apoptotique des cellules tumorales pour un cancer du poumon peut être du à la perte fonctionnelle des protéines issus des gènes suivant :<br> | ||
| Line 360: | Line 363: | ||
<dd><u><i>Cette étude montre une nouvelle fois que le fait d’amener une version wild-type de la p53 au sein d’une population cellulaire p53 mutée induit la mort cellulaire par apoptose.</i></u><br> | <dd><u><i>Cette étude montre une nouvelle fois que le fait d’amener une version wild-type de la p53 au sein d’une population cellulaire p53 mutée induit la mort cellulaire par apoptose.</i></u><br> | ||
| - | |||
| - | + | <span style="float: right">[[Team:SupBiotech-Paris/Ciblage_Cellulaire#drapeau|Haut de page]]</span> | |
| - | + | ||
| - | + | ||
| + | == Conclusion [3,4,5,6,7,8,9] == | ||
| + | Bien que nous n’ayons pu en apporter la preuve par nos propres moyens, de nombreuses études montrent qu’amener une version wild-type d’un gène suppresseur de tumeur au sein d’une cellule tumorale mutée pour ce gène permet le déclenchement de l’apoptose. Des études ''in vivo'' chez l’homme dans le cadre du cancer de la prostate, de l’ovaire et du poumon ont d’ores et déjà été menées et présentent des résultats probants. <br> | ||
| + | La mise en place de cette étude était faite, à l’origine, pour déterminer si l’application du [[Team:SupBiotech-Paris/Introduction1Fr#drapeau|DVS]] dans la lutte anti-cancer du poumon à non petites cellules était viable ou pas. N’ayant pu conclure selon nos propres résultats, c’est l’analyse de diverses publications qui nous a permis de valider la mise en application. Selon ces publications, non seulement la mise en application est confirmée dans le cadre de notre pathologie mais peut désormais être étendue à d’autres cancers comme les carcinomes hépatocellulaires, sur lesquels le fait d’amener un gène suppresseur de tumeur déclenche également le processus d’apoptose. La seule limite étant posée par le tropisme du [[Team:SupBiotech-Paris/Concept1Fr#drapeau|vecteur tissulaire]].<br> | ||
Latest revision as of 01:02, 22 October 2009
Contents |
Le Ciblage cellulaire
Contexte
Après l’action du vecteur tissulaire, viens celle du vecteur cellulaire. Ce dernier est un bactériophage modifié qui a la faculté d’infecter les cellules eucaryotes. Le bactériophage lambda, du fait de sa grande capacité de clonage et une structure de capside adaptée à une présence concentrée de protéines exogènes, est un très bon candidat pour le design d’un vecteur tissulaire eucaryote. La base de penton issue de la capside de l’adénovirus apparait comme un candidat prometteur pour le ciblage du phage lambda. En effet, elle est dotée de plusieurs fonctions telles que la liaison aux récepteurs cellulaires, l’internalisation des particules virales et la libération de la capside par l’endosome.
Objectif
Nos objectifs sont de designer un vecteur cellulaire de type bactériophage Lambda recombiné avec une base de penton issue de l’adénovirus 5 fusionnée à sa protéine D. Le vecteur tissulaire doit être capable d’intégrer la cellule, sortir de l’endosome, transporter son ADN vers le noyau de la cellule et finalement transcrire ce(s) gène(s) thérapeutique(s).
Démarche expérimentale
Dans le cadre du design des gènes du bactériophage recombinant nous avons décidé de fusionner la base de penton de l’adénovirus 5 avec la protéine D du phage lambda. L’extraction de la protéine D à partir du génome du bactériophage Lambda a été menée par réaction de polymérisation en chaine (PCR) avec plusieurs paires de primers. La même stratégie a été prise pour l’extraction de la base de penton de l’adénovirus 5 qui a été extraite d’un plasmide codant pour le virus gracieusement donné par le Dr. Karim Benihoud (UMR8121, CNRS/IGR, Villejuif, France).
Après la formation de la protéine de fusion, celle-ci est introduite dans un plasmide BioBrick. Le plasmide contient une résistance contre un antibiotique pour la confirmation de la transfection du phage recombiné dans la bactérie. Ainsi qu’un gène rapporteur tel que la GFP avec un promoteur eucaryote, le CMV du Simian virus 40 (SV40), pour confirmer la transfection dans les cellules eucaryotes. Cette stratégie nous permet alors de prouver que le bactériophage est capable d’infecter les cellules eucaryotes.
Malheureusement nous n’avons pas été capable de construire la protéine de fusion dans le temps requis. Cependant la littérature scientifique démontre que la confection d’un vecteur tissulaire type bactériophage lambda est possible par fusion de la base de penton de l’adénovirus avec la protéine D (Stefania Piersanti et al. 2004). La séquence centrale de la base de penton, acides aminés 1 à 571, fusionnée avec le bactériophage offre une transfection dans les cellules eucaryotes, tous comme l’utilisation du fragment RGD responsable de l’entrée du virus et la sortie de l’endosome, fragment 286 à 393.
Résultats
Design de la protéine de fusion
Pour le design de la protéine de fusion, nous avons décidé d’extraire séparément la base de penton de l’adénovirus 5 et la protéine D du bactériophage lambda grâce à des primers qui contiennent un site de restriction BalI sur le primer reverse de la protéine D et le primer forward de la base de penton. De plus la protéine de fusion finale contient les fragments spécifiques aux BioBricks à ces deux extrémités.
Pour l’extraction des 2 gènes nous avons utilisé les primers suivants :
Première et deuxième paires pour l’extraction des gènes :
Protéine D du phage Lambda:
Forward : 5' ATG-ACG-AGC-AAA-GAA-ACC-TT 3';
Reverse : 5' AAA-AAA-ATC-CCG-TAA-AAA-AAG-C 3'.
Base de penton de l’adénovirus 5 :
Forward : 5' AAT-GGC-CAA-TGC-GGC-GCG-CGG-CGA-TG 3'
Reverse : 5' CTG-CAG-CGG-CCG-CTA-CTA-GTA-TCA-AAA-AGT-GCG-G 3'
Troisième paire pour l’extention du site de restriction BalI et du préfixe BioBrick pour la protéine D seulement (déjà effectué pour la base de penton).
Forward : 5' CGA-AAA-AAA-TGC-CCT-AAA-AAA-AAC-CGG-T 3'
Reverse : 5' AAT-GGC-CAA-AAA-AAA-TCC-CGT-AAA-AAA-AGC 3'
Quatrième paire pour l’amplification de la protéine de fusion après ligation des deux fragments.
Forward : 5' CTT-AAG-CGC-CGG-CGA-AGA-TC 3'
Reverse : 5' CTG-CAG-CGG-CCG-CTA-CTA-GTA 3'
Les résultats de PCR sont présentés dans la figure 1. Nous observons qu’il y a bien amplification de fragments qui correspondent aux tailles de la base de penton = 1715bp pour l’échantillon 11 et la de protéine D = 385bp pour l’échantillon 7 et 8. Il y a cependant beaucoup de phénomènes de mismatch pendant les cycles d’amplification. Cela pourrait avoir un effet négatif sur le résultat d’amplification final.
Figure 1: PCR des BioBricks de la protéine D (1, 2, 3) et de la base de penton (4, 5, 6), de la protéine D (7 et 8) et de la base de penton (9, 10, 11) avec les sites BalI
Transfection des cellules eucaryotes par le phage lambda recombiné avec la base de penton fusionnée à la protéine D (Stefania Piersanti et al., 2004)
Une étude au par cytofluorimétrie a été faite afin d’analyser le taux de transfection des bactériophages lambda recombinés. La figure 2 montre les résultats de cytofluorimétrie de l’analyse de cellules COS-1 après avoir été exposées à une concentration de 10^6 PFU/cellules de phages recombinants, Pb (1-571) ou Pb (286-393).
Figure 2 : Analyse de la fluorescence de la GFP sur des phages lambda non recombinés (Lambda), des phages lambda recombinés avec le fragment 286-393 de la base de penton (LambdaPb286-393), des phages lambda recombinés avec la base de penton complète (1-571), des adénovirus marqués à la GFP (Ad10 et Ad100)
Premièrement, nous observons que le phage recombiné montre bien une différence de marquage quelque soit le fragment de base de penton utilisé comparé au bactériophage non transformé. Secondement, le phage recombiné avec le fragment RGD seul (286-393) à une fluorescence plus élevée que le phage avec un fragment complet et plus proche de celui des adénovirus (figure 3).
Discussion
Bien que le vecteur tissulaire n’ait pas été fini, la littérature scientifique montre que la création d’un phage recombiné avec une protéine codant la base de penton de l’adénovirus est possible. Il est aussi démontré que les fragments codant pour les séquences RGD seuls ont une plus forte capacité à infecter les cellules eucaryotes comparé au fragment complet de la base de penton (figure 2). Dans le cas de notre application il est alors possible d’utiliser un bactériophage lambda recombiné pour insérer notre gène thérapeutique.
Conclusions
Pour conclure le fragment RGD seul de la base de penton a la meilleure efficacité d’interaction avec les intégrines des cellules eucaryotes. Cependant dans le cadre de notre projet il est plus judicieux d’utiliser la séquence complète de la base de penton (fragment 1-571) car l’utilisation du vecteur cellulaire et du système d’induction par la doxycycline donne une injection très rapide et très ciblée des bactériophages. L’utilisation d’un système de transfection hautement efficace est déconseillé car les phages n’ont pas le temps de se disperser correctement et vont alors infecter plusieurs fois la même cellule. L’utilisation du fragment complet de la base de penton est suffisant pour que le phage infecte correctement les cellules eucaryotes et lui laisse le temps d’avoir une dispersion plus que correct.
Le Plasmide antitumoral
Contexte
Dans le cancer du poumon non à petites cellules, ou NSCLC, comme dans tous cancers, la perte de la capacité apoptotique des cellules tumorales est du à la perte fonctionnelle de divers suppresseurs de tumeur entrant dans la voie de signalisation de la cascade apoptotique.
L’application du DVS dans la lutte anti-cancer repose sur le fait de réactiver cette cascade apoptotique en apportant au sein des cellules tumorales une version wild-type des gènes codant la version saine des suppresseurs de tumeur non-fonctionnels.
C’est le [http://www.sanger.ac.uk/genetics/CGP/cosmic/ projet COSMIC] de [http://www.sanger.ac.uk/ l’institut Sanger] qui nous a permis de déterminer quels gènes apporter au plasmide thérapeutique dans le cadre du cancer du poumon à non petites cellules. Ce projet répertorie en effet toutes les mutations détectées pour chaque type de cancers suivant leur fréquence d’apparition. Ainsi, d’après leurs données, la perte de la capacité apoptotique des cellules tumorales pour un cancer du poumon peut être du à la perte fonctionnelle des protéines issus des gènes suivant :
Ces différents gènes, jouant un rôle prépondérant dans la mise en place du processus apoptotique et étant les plus susceptibles d’avoir mutés dans le cadre d’un cancer du poumon, compose le plasmide thérapeutique.
L’objectif
L’objectif de cette étude est de vérifier si le fait d’amener une version wild-type d’un gène suppresseur de tumeur au sein d’une cellule tumorale pour qui sa version est mutée, induit ou pas le phénomène d’apoptose.
Démarche expérimentale
Lignée cancéreuse et gène apporté
Nous avons sélectionné parmi les lignées cellulaires qui étaient à notre disposition, une lignée cancéreuse dont l’origine cancéreux était du à la mutation d’un gène suppresseur de tumeur. La version wild-type du gène TP53 étant en notre possession, c’est la lignée cancéreuse prostatique p53 muté DU-145 qui retint notre attention.
Nous allons donc tester si le fait d’amener une version wild-type de la protéine p53 (p53wt) au sein de la lignée DU-145 permet le déclenchement du processus d’apoptose.
Protocole de mise en culture :
- Sortir l’ampoule de l’azote liquide
- Placer l’ampoule dans un bain-marie à 37°C pendant 5 minutes
- Dans un falcon 50 ml, mettre 9 ml de MEM 10% + 1 ml d’ampoule
- Centrifuger 5 min à 1200 rpm
- Aspirer le surnageant sans toucher aux cellules culotées (élimination du DMSO)
- Resuspendre le culot dans 1 ml de milieu
- Déposer le tout dans une nouvelle flasque T25 contenant 5 ml de milieu
- Incubation à 37°C
- Ne pas oublier de changer le milieu le lendemain pour éliminer les traces de DMSO
- Après une semaine, les cellules sont à confluence 100%
Incorporation du gène TP53
L’incorporation du plasmide contenant p53wt, pcDNA3 CMV+p53wt, au sein des cellules DU-145 s’est effectuée par électroporation.
Matériel :
- Cellules DU-145
- Plasmide pcDNA3 CMV+p53wt
- Milieu de culture électrocompétent
- Trypsine
- PBS
- Bac à glace
- Cuvette d’électrotransfert
- Centrifugeuse
- Incubateur
- Electroporateur (cliniporateur)
Protocole:
- Aspirer le milieu du T25 contant les DU-145
- Rincer au PBS
- Déposer 500 µl de trypsine et laisser agir 3 minutes à température ambiante
- Ajouter 5 ml de MEM 10% pour neutraliser la trypsine
- Suspendre les cellules
- Récupérer le milieu contenant les DU-145 dans un tube et centrifuger à 1000rpm pendant 10 minutes
- Aspirer le surnageant et resuspendre le culot dans Xµl (X= 90µl x Nombre de cuves) de milieu électrocompétent (environ 5x105 cellules par cuves)
- Suspendre votre solution d’ADN dans du milieu électrocompétent (18x10-2g/L)
- Ajouter 10µl de solution d’ADN par cuve
- Ajouter 90µl de la suspension cellulaire
- Mettre les cuves dans la glace
- Passer les cuves à l’électroporateur (cliniporateur) et enregistrer chaque résultat
- Incuber les cuves à 37°C pendant 30 minutes
- Mettre le contenu de chaque cuve dans un tube stérile, ajouter 3ml de milieu de culture MEM 10%, puis incuber à 37°C pendant le temps nécessaire (jusqu’au test à l’annexine V)
Détection de l’apoptose
La détection des cellules apoptotiques s’est effectuée par le test à l’annexine V :
En phase précoce de l’apoptose, on observe la translocation de la phosphatidyl-sérine à l’extérieur de la membrane plasmique. Celle-ci est mise en évidence par fixation spécifique de l'annexine V couplée à un fluorophore et analysée par cytométrie en flux.
Matériel :
- Iodure de propidium 1 mg/ml In vitrogen conservé au frigidaire à diluer 10 fois
- Annexine V
- Tampon annexine
Travailler le plus possible dans l’obscurité (fluorophore photolabile)
Protocole :
- Récupérer le milieu de culture (3 ml), le déposer dans un falcon 50 ml
- Rincer la culture avec 3 ml de PBS, les déposer dans le falcon
- Décoller les cellules à la trypsine, les déposer dans le falcon
- Centrifuger
- Reprendre le culot dans 0.5 ou 1 ml de PBS froid en fonction du niveau de confluence
- Prélever 10 µl pour un comptage et centrifuger
- Re-suspendre le culot dans du tampon annexine à la concentration de 1*106 cellule/ml
- Pipetter 2 aliquots de 100 µl dans 2 tubes FACS
- Ajouter dans chaque tube 5 µl d’annexine V et 1 µl de iodure de propidium
- Incuber 15 min à RT
- Arrêter la réaction en plaçant les tubes dans la glace fondante
- Ajouter 400 µl de tampon d’annexine V
- Lire au FACS le plus rapidement possible en conservant les tubes dans la glace
Déroulement de l’étude
Ne connaissant pas le temps d’expression du plasmide au sein de la lignée DU-145, nous avons réalisé un suivi cinétique de l’induction de l’apoptose en pratiquant un test à l’annexine V toutes les 6 heures pendant 48h après son électroporation. De ce fait, en couplant les taux d’apoptose de la population témoin (électroporation à vide) et de la population test (électroporation avec plasmide) avec leur taux de croissance respectifs, nous serons en mesure de déterminer l’impacte de p53wt sur l’induction de l’apoptose. La population témoin permettant d’éliminer les morts cellulaires dus à l’électroporation et au transfert de culture.
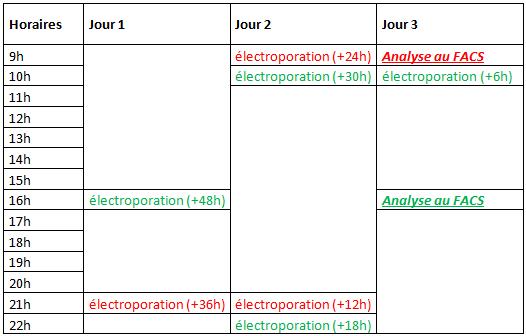
N’ayant pas eu un accès continu au cytomètre en flux, nous avons regroupé l’ensemble des 48h d’analyse en deux runs de cytométrie. Chaque créneau horaire de l’étude est représenté par une population cellulaire distincte. Ainsi nous avons réalisé 14 électroporations correspondant aux 7 créneaux horaires : +6h, +12h, +18h, +24h, +30h, +36h et +48h (deux par créneaux : population test + population témoin).
Voici le planning de répartition des électroporations:
Trois populations cellulaires ont donc été respectivement électroporées 12h, 24h et 36h avant le premier run de cytométrie (en rouge, à 9h, jour 3), quatre autres 6h, 18h, 30h et 48h avant le second run (en vert, à 16h, jour 3).
La première analyse cytométrique nous a permis d’obtenir les données pour le suivi à +12h, +24h et +36h, tandis que la seconde, nous a permis d’obtenir les données pour le suivi à +6h, 18h, +30h et +48h.
En couplant toutes ces données, on obtient un suivi sur 48h de l’induction de l’apoptose après électroporation de p53wt.
Résultats [1,2]
Chaque population cellulaire, représentant les différentes tranches horaires du suivi, a subi un test à l’annexine V à l’instant escompté. Malheureusement, une mauvaise dilution du tampon de l’annexine a causé la mort de toutes les populations cellulaires lors du test. Bien que les résultats furent probants pour les suivis à +24h, +30h et +48h par simple comparaison des populations contrôles et tests au microscope (figure 1), nous n’avons pu le confirmer par l’analyse cytométrique.
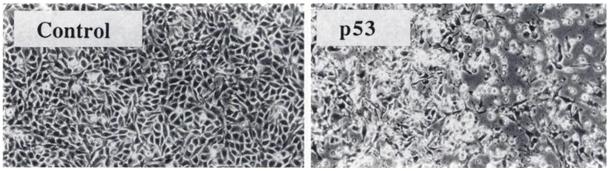
Figure 1 : morphologie des cellules avec ou sans incorporation de p53 wild-type
N’ayant pu commencer la culture des DU-145 que début octobre, les deux semaines qui nous a fallu pour atteindre la confluence nécessaire à l’expérimentation n’ont pas laissé place à la pratique d’un second essai…
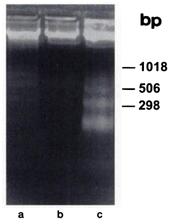
Cependant, de nombreuses études ont montré que le fait d’amener p53 wild type au sein de cellules tumorales p53 mutées déclenchait le processus d’apoptose. C’est le cas notamment de l’étude menée par Chunlin Yang en 1995 qui a travaillé, tout comme nous, sur des cellules cancéreuses prostatiques p53 mutées (Tsu-pr1). La transfection de p53 wild type n’a pas été réalisée par électroporation mais en infectant les cellules tumorales avec des adénovirus non réplicatifs contenant p53wt (AdCMV.p53). Quarante-huit heures après avoir infecté une population tumorale avec AdCMV.p53, une forte expression de p53 est corrélée avec un taux important de mort cellulaire. Si les populations témoins (cellules non-infectées et cellules infectées avec des adénovirus contenant le gène LacZ, AdCMV.NLSßgal) montrent une morphologie tout à fait similaire et saine, une condensation et un détachement cellulaire sont observés chez la population p53 infectée. Afin de vérifier si le processus de mort suivi par ces cellules correspond bien à la voie apoptotique, une migration sur gel d’agarose de leur génome a été réalisée.
Figure 2 : électrophorèse sur gel d’agarose d’ADN isolé de cellules non-infectées (a), infectées par AdCMV.NLSßgal (b) et AdCMV.p53 (c).
Les cellules infectées par AdCMV.p53 montrent une multitude de bandes (laddering pattern) tandis que les cellules non-infectées ou infectées par AdCMV.NLSßgal n’en montrent qu’une seul et unique de haut poids moléculaire. Ces résultats indiquent que la mort cellulaire induite par p53 wild type est d’origine apoptotique avec l’observation de la fragmentation du génome, conséquence de l’activité de la CAD (Caspase Activated DNase), une endonucléase spécifique au processus d’apoptose.
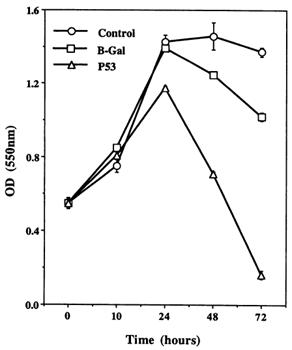
Un test MTT à permit de quantifier l’effet induit par l’expression de p53 wild type chez les cellules infectées.
Figure 3 : effet de l’AdCMV.p53 sur la survie cellulaire. Les cellules témoins et celles infectées à l’AdCMV.p53 ont été incubé dans du milieu serum-free après 1h d’infection.
En l’absence de sérum, les cellules non-infectées et ßgal infectées continuent de proliférer. En revanche, pour les cellules p53 infectées, la prolifération est stoppée et suivie d’une importante chute de la population. Après 72h, la quasi-totalité des cellules p53 infectées sont mortes (figure 3).
Selon cette étude, il apparait clairement que le fait d’amener une version wild-type de la p53 au sein d’une population cellulaire p53 mutée induit le phénomène d’apoptose et réduit de manière significative la population tumorale.
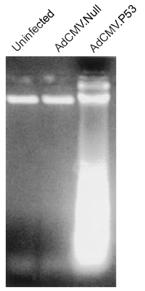
Des résultats similaires ont été rapportés par l’étude menée par Corrado Cirielli (en 1999) mais portant cette fois-ci sur la lignée cancéreuse U251 issue d’un gliome. Les mêmes types d’analyses que celles réalisées au cours de l’étude précédente ont été pratiquées.
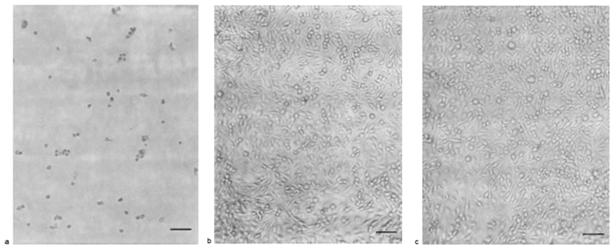
Figure 4 : morphologie des cellules infectées par AdCMV.p53 (a), non-infectées (b) ou infectées par AdCMV.NULL (c), une semaine après infection.
Les populations témoins (b et c) prolifèrent et forment un tapis cellulaire une semaine après le début de l’expérience tandis que la population test (a) montrent très peu de cellules adhérentes (perte cellulaire importante) et un changement morphologique conséquent : les cellules sont sphériques.
Figure 5 : électrophorèse sur gel d’agarose d’ADN isolé de cellules non-infectées, infectées par AdCMV.NULL et AdCMV.p53.
Après infection à l’AdCMV.p53, les cellules U-251 montrent une fragmentation de leurs génomes caractéristique du processus d’apoptose.
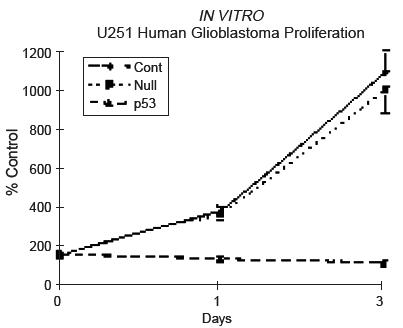
Figure 6 : prolifération des populations témoins (non-infectées ou AdCMV.NULL infectées) et de la population test par suivi de la densité optique après un test MTT.
Conclusion [3,4,5,6,7,8,9]
Bien que nous n’ayons pu en apporter la preuve par nos propres moyens, de nombreuses études montrent qu’amener une version wild-type d’un gène suppresseur de tumeur au sein d’une cellule tumorale mutée pour ce gène permet le déclenchement de l’apoptose. Des études in vivo chez l’homme dans le cadre du cancer de la prostate, de l’ovaire et du poumon ont d’ores et déjà été menées et présentent des résultats probants.
La mise en place de cette étude était faite, à l’origine, pour déterminer si l’application du DVS dans la lutte anti-cancer du poumon à non petites cellules était viable ou pas. N’ayant pu conclure selon nos propres résultats, c’est l’analyse de diverses publications qui nous a permis de valider la mise en application. Selon ces publications, non seulement la mise en application est confirmée dans le cadre de notre pathologie mais peut désormais être étendue à d’autres cancers comme les carcinomes hépatocellulaires, sur lesquels le fait d’amener un gène suppresseur de tumeur déclenche également le processus d’apoptose. La seule limite étant posée par le tropisme du vecteur tissulaire.
 "
"