Team:SupBiotech-Paris/Matériel & Méthode
From 2009.igem.org
Enguerrand (Talk | contribs) (New page: {{Template:Supbiotechcss.css}} {{Template:SupbiotechparisFr}}) |
|||
| (28 intermediate revisions not shown) | |||
| Line 1: | Line 1: | ||
| - | {{Template: | + | {{Template:Supbiotechcss12.css}} |
{{Template:SupbiotechparisFr}} | {{Template:SupbiotechparisFr}} | ||
| + | <html> | ||
| + | <div style="float: right; margin-right: -85px;"> | ||
| + | <a href="https://2009.igem.org/Team:SupBiotech-Paris/Bibliographie#drapeau" target="_self"> | ||
| + | <img title="On passe à la page suivante !" style="width: 100px;" src="https://static.igem.org/mediawiki/2009/e/e9/Suivant.png";> | ||
| + | </a></div> | ||
| + | </html> | ||
| + | == Electrophorèse == | ||
| + | |||
| + | === Matériel === | ||
| + | |||
| + | Template d’ADN<br> | ||
| + | Tampon de charge<br> | ||
| + | Marqueur de taille moléculaire<br> | ||
| + | Tampon TAE 1X<br> | ||
| + | Bromure d’éthidium (BET) <br> | ||
| + | Agarose<br> | ||
| + | Matériel de migration et de détection pour électrophorèse en gel d’agarose<br> | ||
| + | |||
| + | === Procédure === | ||
| + | |||
| + | 1. Préparation du gel d’agarose : <br> | ||
| + | a) Pour les bio-briques : <br> | ||
| + | Préparer un gel d’agarose à 2% (1,5 gramme d’agarose dans 100mL d’H2O bidistillée + 15µL de BET). <br> | ||
| + | b) Pour isoler l’ADN du phage lambda : <br> | ||
| + | Préparer un gel d’agarose à 0,7% (0,4 gramme d’agarose dans 100mL d’H2O bidistillée + 15µL de BET). <br> | ||
| + | 2. Mettre l’agarose à chauffer aux micro-ondes 5 à 10 minutes. <br> | ||
| + | 3. Préparer la plaque d’électrophorèse. <br> | ||
| + | Faire bien attention à l’étanchéité de la plaque. <br> | ||
| + | 4. Ajouter le tampon TAE 1X jusqu’à recouvrir le gel d’agarose. <br> | ||
| + | 5. Préparer les échantillons. <br> | ||
| + | Mélanger 5µL de tampon de charge pour 15µL d’échantillon d’ADN. <br> | ||
| + | Préparer le marqueur de taille moléculaire si besoin : <br> | ||
| + | High range : 0,5µL d’ADN + 1µL Tampon de charge du marqueur + 4,5µL d’H2O bi distillée. <br> | ||
| + | 6. Vortexer puis centrifuger 2 secondes. <br> | ||
| + | 7. Déposer délicatement les échantillons et le marqueur de masse moléculaire. <br> | ||
| + | Ne pas utiliser les puits situé sur les coté du gel. <br> | ||
| + | Déposer le marqueur de masse moléculaire au centre du gel pour faciliter l’analyse de la taille des échantillons. <br> | ||
| + | Une plaque 8 puits a une contenance d’environ 45µL d’échantillon. <br> | ||
| + | Une plaque 15 puits peut contenir 15µL d’échantillon. <br> | ||
| + | 7. Placer les électrodes de la cuve d’électrophorèse en sorte que l’ADN migre de l’anode vers la cathode. <br> | ||
| + | 8. Déclencher la migration du gel de 85 volts à 130 volts pendant 45 à 80 minutes. <br> | ||
| + | Vérifier qu’il y a bien apparition de bulles due au courant électrique et que l’ADN migre dans le bon sens. <br> | ||
| + | |||
| + | <span style="float: right">[[Team:SupBiotech-Paris/Matériel & Méthode#drapeau|Haut de page]]</span> | ||
| + | |||
| + | == Extraction d’ADN à partir d’un gel d’agarose (Nucleospin Clontech) == | ||
| + | === Matériel === | ||
| + | 30ml de tampon NT<br> | ||
| + | 14ml de tampon NT3<br> | ||
| + | 15ml de tampon NE<br> | ||
| + | 50 colonnes Nucleospin Extract II<br> | ||
| + | 50 tubes collecteurs Nucleospin (2ml) <br> | ||
| + | Scalpel stérile<br> | ||
| + | Balance<br> | ||
| + | Vortex<br> | ||
| + | Incubateur (50°C) <br> | ||
| + | Centrifugeuse (11000g) <br> | ||
| + | Tubes 1.5ml stérile<br> | ||
| + | |||
| + | === Procédure === | ||
| + | 1. Exciser un fragment du gel d’agarose contenant le fragment d’ADN, avec le scalpel, en prenant soin de réduire au minimum le volume de gel. <br> | ||
| + | 2. Déterminer le poids de la tranche de gel et la transférer à un tube stérile. <br> | ||
| + | 3. Pour chaque 100 mg gel d'agarose ajouter 200μl de tampon NT. <br> | ||
| + | Pour les gels contenant plus de 2% d'agarose, doubler du volume de tampon NT. <br> | ||
| + | Le poids maximal par tranche de gel, par NucleoSpin® Extract II, est de 400 mg ou 200 mg pour les pourcentages élevés (> 2%), dans ce cas, 2 étapes sont nécessaires (étapes de liaison de l’ADN : 6 et 7). <br> | ||
| + | 4. Incuber les échantillons à 50 ° C jusqu'à ce que le gel soit dissout (5-10 min). <br> | ||
| + | 5. Vortexer l'échantillon brièvement tous les 2 à 3 min jusqu'à ce que le gel soit complètement dissout. <br> | ||
| + | 6. Placer une colonne NucleoSpin ® Extrait II dans un tube collecteur Nucleospin de 2 ml et mettre l'échantillon, puis centrifuger pendant 1 min à 11000g. <br> | ||
| + | 7. Jeter la solution contenue dans le tube collecteur Nucleospin et replacer la colonne NucleoSpin Extract II ® dans le même tube collecteur Nucleospin. <br> | ||
| + | 8. Ajouter 600μl de tampon NT3 et centrifuger pendant 1 min à 11000g. <br> | ||
| + | 9. Jeter la solution contenue dans le tube collecteur Nucleospin et replacer la colonne NucleoSpin Extract II ® dans le tube collecteur Nucleospin. <br> | ||
| + | 10. Centrifuger pendant 2 min à 11000g pour supprimer le tampon NT3. <br> | ||
| + | Assurez-vous que la colonne Nucleospin ne soit pas en contact avec la solution éluée lors de la centrifugation (dans le tube collecteur Nucleospin). <br> | ||
| + | L'éthanol résiduel issu du tampon NT3 inhibe les réactions ultérieures et doit être retiré à cette étape. En plus de la centrifugation, une suppression totale peut être réalisée à partir d’une incubation de la colonne NucleoSpin ® Extrait II pendant 2-5 min à 70°C avant l'élution. <br> | ||
| + | 11. Placer la colonne NucleoSpin ® Extract II dans un tube propre de 1,5 ml. <br> | ||
| + | 12. Ajouter 15-50μl de tampon d'élution NE, puis incuber à température ambiante pendant 1 min pour augmenter le rendement de l'ADN élué. <br> | ||
| + | 13. Centrifuger pendant 1 min à 11000g. <br> | ||
| + | Le rendement des grands fragments (> 5-10 kb) peut être augmenté par l'utilisation de tampon d'élution préchauffé (70 ° C): Pour l'élution, ajouter des tampons d'élution préchauffée et incuber à température ambiante pendant 1 min avant de recueillir l'éluant par centrifugation. <br> | ||
| + | |||
| + | <span style="float: right">[[Team:SupBiotech-Paris/Matériel & Méthode#drapeau|Haut de page]]</span> | ||
| + | |||
| + | == Préparation d’Escherichia coli électrocompétentes == | ||
| + | === Matériel === | ||
| + | 500µl de culture d’E.Coli dans du milieu de culture LB<br> | ||
| + | Milieu de culture LB<br> | ||
| + | 60ml de glycérol à 10%<br> | ||
| + | Ethanol<br> | ||
| + | Incubateur avec rotation<br> | ||
| + | Bac de Glace<br> | ||
| + | Tubes de 1.5ml<br> | ||
| + | Tubes Falcon 50ml<br> | ||
| + | Tubes pour centrifugeuse 300ml<br> | ||
| + | Centrifugeuse réfrigérée à 4°C<br> | ||
| + | |||
| + | === Procédure === | ||
| + | 0. Préparer une pré-culture d’E.Coli dans 5ml de milieu de culture LB (toute la nuit à 37°C avec rotation). <br> | ||
| + | Préparer la centrifugeuse à 4°C<br> | ||
| + | 1. Mettre la pré-culture d’E.Coli dans 450ml de milieu de culture LB et incuber à 37°C avec agitation pendant 3 heures. <br> | ||
| + | 2. Placer 225ml de la solution dans 2 tubes pour centrifugeuse. <br> | ||
| + | 3. Placer les deux tubes dans la glace pendant 15 minutes. <br> | ||
| + | 4. Centrifuger à 2000g pendant 10 minutes à 4°C. <br> | ||
| + | 7. Eliminer le surnageant et remettre délicatement en suspension avec 200ml d’eau froide stérile. <br> | ||
| + | Ajouter d’abord 10 à 20ml d’eau froide stérile et resuspendre par pipetage, puis ajouter le reste des 200ml. <br> | ||
| + | 8. Centrifuger à 2000g pendant 10 minutes à 4°C. <br> | ||
| + | 9. Eliminer le surnageant et remettre délicatement en suspension avec 200ml d’eau froide stérile. | ||
| + | Ajouter d’abord 10 à 20ml d’eau froide stérile et resuspendre par pipetage, puis ajouter le reste des 200ml. <br> | ||
| + | 10. Placer les tubes dans la glace pendant 30 minutes. <br> | ||
| + | 11. Centrifuger à 2000g pendant 15 minutes à 4°C. <br> | ||
| + | 12. Eliminer le surnageant et remettre délicatement en suspension avec 25ml de glycérol à 10%.<br> | ||
| + | On peut éventuellement transférer la solution dans un tube Falcon de 50ml. <br> | ||
| + | 13. Placer les tubes dans la glace pendant 30 minutes. <br> | ||
| + | 14. Centrifuger à 1500g pendant 15 minutes à 4°C. <br> | ||
| + | 15. Eliminer le surnageant et ajouter 500µl de glycérol à 10%.<br> | ||
| + | 16. Resuspendre les cellules dans un volume total de 1ml. <br> | ||
| + | 17. Aliquoter 50µl par tube (tubes déjà dans la glace). <br> | ||
| + | 18. Stocker à -80°C<br> | ||
| + | |||
| + | <span style="float: right">[[Team:SupBiotech-Paris/Matériel & Méthode#drapeau|Haut de page]]</span> | ||
| + | |||
| + | == Electroporation d’Escherichia coli électrocompétentes == | ||
| + | === Matériel === | ||
| + | Milieu LB liquide<br> | ||
| + | Milieu LB solide (milieu de culture LB + agarose + antibiotique adapté) <br> | ||
| + | Bactéries Escherichia coli électrocompétentes congelées (40 microlitres dans un tube eppendorf)<br> | ||
| + | ADN<br> | ||
| + | Bac rempli de glace<br> | ||
| + | Cuvettes d’électroporation (2 mm) <br> | ||
| + | Electroporateur<br> | ||
| + | Etuve avec agitation<br> | ||
| + | |||
| + | === Procédure === | ||
| + | 0. Placer la cuvette d’électroporation dans de la glace. <br> | ||
| + | 1. Mettre le tube contenant les bactéries (environ 40 microlitres) à température ambiante, afin de dégeler le milieu. Le placer ensuite dans de la glace. <br> | ||
| + | 2. Régler l’électroporateur avec les paramètres suivants : <br> | ||
| + | <div style="margin-left: 100px;"> | ||
| + | Voltage 2500 V<br> | ||
| + | Capacitance 25 µF<br> | ||
| + | Résistance 200 Ω<br> | ||
| + | Cuvette 2 mm<br> | ||
| + | </div> | ||
| + | 3. Ajouter un microlitre de la solution d’ADN au tube contenant les bactéries. <br> | ||
| + | 4. Placer le tube une minute dans la glace. <br> | ||
| + | 5. Transférer le contenu du tube dans la cuvette d’électroporation. <br> | ||
| + | 6. Sécher l’extérieur de la cuvette avec du papier absorbant. La mettre dans le compartiment prévu de l’électroporateur. <br> | ||
| + | 7. Déclencher le pulse.<br> | ||
| + | 8. Rajouter immédiatement 1mL de milieu de culture dans la cuvette. <br> | ||
| + | Attention : un retard de 3 minutes diminue l’efficacité de la transformation de 90%.<br> | ||
| + | 9. Transférer le contenu de la cuvette dans un tube. Le mettre à incuber une heure à 37°C sous agitation. <br> | ||
| + | Tremper les cuvettes quelques minutes dans de l’eau de javel. Les rincer abondamment avec de l’eau du robinet, puis avec de l’eau bidistillée et de l’éthanol à 70% (pas d’acétone), avant de les laisser sécher. Elles pourront ainsi être réutilisées. <br> | ||
| + | 10. Mettre les bactéries en culture sur un milieu LB solide. <br> | ||
| + | |||
| + | <span style="float: right">[[Team:SupBiotech-Paris/Matériel & Méthode#drapeau|Haut de page]]</span> | ||
| + | |||
| + | == Préparation de Mycobacterium électrocompétentes == | ||
| + | === Matériel === | ||
| + | 500μl de Culture de Mycobacterium à -80°C<br> | ||
| + | Glycérol 10%<br> | ||
| + | Tubes de 1.5ml<br> | ||
| + | Centrifugeuse<br> | ||
| + | |||
| + | === Procédure === | ||
| + | 0. Toute la manipulation se déroule et tous les milieux sont à température ambiante. <br> | ||
| + | 1. Transférer les cellules de votre échantillon congelé dans des tubes de centrifugation. <br> | ||
| + | 2. Centrifuger les cellules à 3600rpm pendant 10 min, puis éliminer le surnageant. <br> | ||
| + | 3. Laver les cellules avec 1/2 volume (25ml) du glycérol 10% stérile en pipetant doucement jusqu’à ce que la masse de cellules soit dissoute. <br> | ||
| + | 4. Laver et culotter les cellules comme l’étape 2 et 3; avec 1/4 vol (12.5ml) de glycérol 10% stérile. <br> | ||
| + | 5. Laver et culotter les cellules comme l’étape 2 et 3; avec 1/8 vol (6,75ml) de glycérol 10% stérile. <br> | ||
| + | 6. Laver et culotter les cellules comme l’étape 2 et 3; avec 1/10 vol (5ml) de 10% glycérol stérile. <br> | ||
| + | 7. Laver et culotter les cellules comme l’étape 2 et 3; avec 1/25 vol (2ml) de 10% glycérol stérile. <br> | ||
| + | 8. Aliquoter 100μL de cellules dans des tubes de 1,5ml. <br> | ||
| + | 9. Laisser refroidir rapidement dans un bain de glace.<br> | ||
| + | 10. Puis stocker à -80°C, ou utiliser immédiatement (efficacité de l’électrotransfert augmentée). <br> | ||
| + | |||
| + | <span style="float: right">[[Team:SupBiotech-Paris/Matériel & Méthode#drapeau|Haut de page]]</span> | ||
| + | |||
| + | == Electroporation de Mycobacterium électrocompétentes == | ||
| + | === Matériel === | ||
| + | Milieu Loewenstein liquide <br> | ||
| + | Milieu Loewenstein solide (milieu de culture Loewenstein coagulé + antibiotique adapté) <br> | ||
| + | Mycobacterium électrocompétentes congelées (40 microlitres dans un tube) <br> | ||
| + | ADN<br> | ||
| + | Bac rempli de glace<br> | ||
| + | Cuvettes d’électroporation (2 mm) <br> | ||
| + | Electroporateur<br> | ||
| + | Etuve avec agitation<br> | ||
| + | |||
| + | === Procédure === | ||
| + | 0. Placer la cuvette d’électroporation dans de la glace. <br> | ||
| + | 1. Mettre le tube contenant les bactéries (environ 40 microlitres) à température ambiante afin de dégeler le milieu. Le placer ensuite dans de la glace. <br> | ||
| + | 2. Régler l’électroporateur avec les paramètres suivants : <br> | ||
| + | <div style="margin-left: 100px;"> | ||
| + | Voltage 2500 V<br> | ||
| + | Capacitance 25 µF<br> | ||
| + | Résistance 1000 Ω<br> | ||
| + | Cuvette 2 mm<br> | ||
| + | </div> | ||
| + | 3. Ajouter un microlitre de la solution d’ADN au tube contenant les bactéries. <br> | ||
| + | 4. Placer le tube une minute dans la glace. <br> | ||
| + | 5. Transférer le contenu du tube dans la cuvette d’électroporation. <br> | ||
| + | 6. Sécher l’extérieur de la cuvette avec du papier absorbant. La mettre dans le compartiment prévu de l’électroporateur. <br> | ||
| + | 7. Déclencher le pulse. <br> | ||
| + | 8. Rajouter immédiatement 1mL de milieu de culture dans la cuvette. <br> | ||
| + | Attention : un retard de 3 minutes diminue l’efficacité de la transformation de 90%.<br> | ||
| + | 9. Transférer le contenu de la cuvette dans un tube. Le mettre à incuber une heure à 37°C sous agitation. <br> | ||
| + | Tremper les cuvettes quelques minutes dans de l’eau de javel. Les rincer abondamment avec de l’eau du robinet, puis avec de l’eau bidistillée et de l’éthanol à 70% (pas d’acétone), avant de les laisser sécher. Elles pourront ainsi être réutilisées. <br> | ||
| + | 10. Mettre les bactéries en culture sur un milieu Loewenstein solide. <br> | ||
| + | |||
| + | <span style="float: right">[[Team:SupBiotech-Paris/Matériel & Méthode#drapeau|Haut de page]]</span> | ||
| + | |||
| + | == Restriction == | ||
| + | === Matériel === | ||
| + | 10µL d’ADN (1µL pour 0,5µg d'ADN) <br> | ||
| + | 3µL de tampon de restriction (1X du volume total) <br> | ||
| + | 1µL d’enzyme de restriction<br> | ||
| + | 3µL BSA (1X du volume total) <br> | ||
| + | 15µL d’eau distillée stérile<br> | ||
| + | Volume Total 30µL<br> | ||
| + | Tubes de 1.5ml<br> | ||
| + | |||
| + | === Procédure === | ||
| + | 1. Dans un tube eppendorf stérile. <br> | ||
| + | 2. Mettre Tampon 3µL de tampon. <br> | ||
| + | 3. Ajouter l'ADN. <br> | ||
| + | 4. Ajouter 3µL de BSA. <br> | ||
| + | 5. Puis, ajouter 1µL d’enzymes de restriction. <br> | ||
| + | 6. Et compléter avec l’eau distillée stérile. <br> | ||
| + | VOLUME TOTAL = 30µL. <br> | ||
| + | 7. Mettre dans un thermocycler avec les conditions: <br> | ||
| + | <div style="margin-left: 100px;"> | ||
| + | a. 1 - 2 H à 37°C, <br> | ||
| + | b. 20 min à 80°C pour l'inactivation des enzymes, <br> | ||
| + | c. Laisser à 4°C jusqu'à ouverture du thermocycler. <br> | ||
| + | </div> | ||
| + | NOTE : si on peut laisser plus de 6h d'incubation pour la restriction, laisser plus longtemps pour avoir une restriction de meilleure qualité! <br> | ||
| + | |||
| + | <span style="float: right">[[Team:SupBiotech-Paris/Matériel & Méthode#drapeau|Haut de page]]</span> | ||
| + | |||
| + | == Linéarisation == | ||
| + | === Matériel === | ||
| + | Template d’ADN<br> | ||
| + | 1µl de phosphatase alcaline <br> | ||
| + | 3µl de tampon de phosphatase alcaline<br> | ||
| + | Eau Pure Volume total 30µl<br> | ||
| + | Bain marie ou plaque chauffante<br> | ||
| + | Tubes de 1.5ml<br> | ||
| + | |||
| + | === Procédure === | ||
| + | 0. Préparer un bain marie à 37°C.<br> | ||
| + | 1. Ajouter le tampon de la phosphatase alcaline concentration 1X dans le template d’ADN, soit 3µL de tampon pour 30µL de volume final. <br> | ||
| + | 2. Ajouter 1µL d’alkaline phosphatase. <br> | ||
| + | 3. Incuber 1 heure à 37°C. <br> | ||
| + | 4. Inactivation par la chaleur de la phosphatase alcaline 5 minutes à 65°C. <br> | ||
| + | Procéder à la ligation juste après la linéarisation. <br> | ||
| + | |||
| + | <span style="float: right">[[Team:SupBiotech-Paris/Matériel & Méthode#drapeau|Haut de page]]</span> | ||
| + | |||
| + | == Ligation == | ||
| + | === Matériel === | ||
| + | 3µl de tampon de ligation 10X<br> | ||
| + | 1µl de T4 DNA Ligase<br> | ||
| + | 100 à 1000µg en solution d’insert d’ADN<br> | ||
| + | 1µl de vecteur d’ADN à 0.05g/l<br> | ||
| + | Eau stérile pour un volume final de 30µl<br> | ||
| + | Tubes de 1.5ml<br> | ||
| + | |||
| + | === Procédure === | ||
| + | 1. Dans un tube de 1,5ml, mettre 2µl de tampon de ligation. <br> | ||
| + | 2. Ajouter l’insert d’ADN ainsi que 1µl de vecteur. <br> | ||
| + | 3. Ajouter 1µl de T4 DNA Ligase. <br> | ||
| + | 4. Compléter avec de l’eau stérile pour avoir 30µl de volume final. <br> | ||
| + | 5. Laisser agir pendant 35 minutes à 22.5°C. <br> | ||
| + | 6. Dénaturer la T4 DNA Ligase à 65°C pendant 10 minutes.<br> | ||
| + | |||
| + | <span style="float: right">[[Team:SupBiotech-Paris/Matériel & Méthode#drapeau|Haut de page]]</span> | ||
| + | |||
| + | == PCR (Kit Promega) == | ||
| + | === Matériel === | ||
| + | Template d’ADN<br> | ||
| + | Primer 5’ à 3’<br> | ||
| + | Primer 3’ à 5’<br> | ||
| + | GoTaq Hot Start Green Mix (Promega) <br> | ||
| + | Thermocycler<br> | ||
| + | |||
| + | === Procédure === | ||
| + | 1. Mettre le kit GoTaq Hot Start Green mix à température ambiante, vortexer puis centrifuger brièvement pour concentrer le mix au fond du tube. <br> | ||
| + | |||
| + | 2. Préparer le mix de PCR suivant : <br> | ||
| + | |||
| + | |||
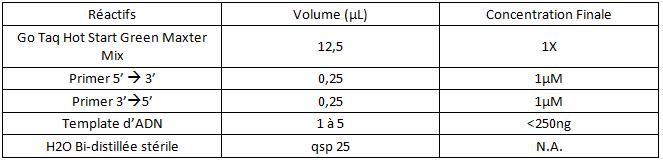
| + | a. Pour 25µL : <br> | ||
| + | |||
| + | [[Image:PCR25.jpg]]<br> | ||
| + | |||
| + | |||
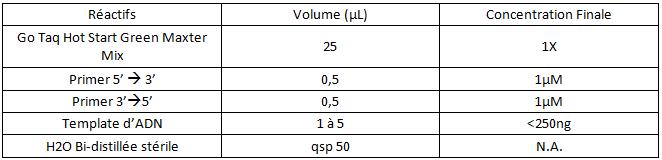
| + | b. Pour 50µL : <br> | ||
| + | |||
| + | [[Image:PCR50.jpg]]<br> | ||
| + | |||
| + | |||
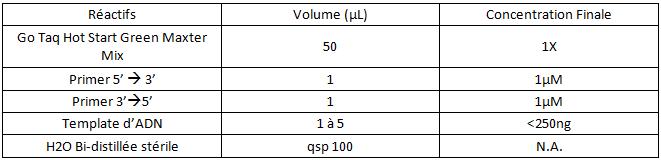
| + | c. Pour 100µL : <br> | ||
| + | |||
| + | [[Image:PCR100.jpg]]<br> | ||
| + | |||
| + | |||
| + | 3. Ajouter dans l'ordre donné dans les tableaux. <br> | ||
| + | |||
| + | Faire attention à bien insérer les volumes de primers, de template sur les bords du tube afin de voir tous les volumes déposés. <br> | ||
| + | |||
| + | 4. Vortexer le volume total<br> | ||
| + | |||
| + | 5. Centrifuger "Start - stop" (2 à 5 secondes) <br> | ||
| + | |||
| + | Si le thermocycler n'a pas de couvercle chauffant ajouter 1 à 2 gouttes d'huile minérale stérile. <br> | ||
| + | |||
| + | 6. Insérer la préparation dans le thermocycler, puis lancer les cycles<br> | ||
| + | |||
| + | Cycles : <br> | ||
| + | [[Image:PCRcycle.jpg]]<br> | ||
| + | |||
| + | |||
| + | 7. Vérifier vos produits de PCR par électrophorèse. <br> | ||
| + | |||
| + | |||
| + | <span style="float: right">[[Team:SupBiotech-Paris/Matériel & Méthode#drapeau|Haut de page]]</span> | ||
| + | |||
| + | |||
| + | <html> | ||
| + | <div style="float: right; margin-right: -85px;"> | ||
| + | <a href="https://2009.igem.org/Team:SupBiotech-Paris/Bibliographie#drapeau" target="_self"> | ||
| + | <img title="On passe à la page suivante !" style="width: 100px;" src="https://static.igem.org/mediawiki/2009/e/e9/Suivant.png";> | ||
| + | </a></div> | ||
| + | </html> | ||
Latest revision as of 02:29, 21 October 2009
Electrophorèse
Matériel
Template d’ADN
Tampon de charge
Marqueur de taille moléculaire
Tampon TAE 1X
Bromure d’éthidium (BET)
Agarose
Matériel de migration et de détection pour électrophorèse en gel d’agarose
Procédure
1. Préparation du gel d’agarose :
a) Pour les bio-briques :
Préparer un gel d’agarose à 2% (1,5 gramme d’agarose dans 100mL d’H2O bidistillée + 15µL de BET).
b) Pour isoler l’ADN du phage lambda :
Préparer un gel d’agarose à 0,7% (0,4 gramme d’agarose dans 100mL d’H2O bidistillée + 15µL de BET).
2. Mettre l’agarose à chauffer aux micro-ondes 5 à 10 minutes.
3. Préparer la plaque d’électrophorèse.
Faire bien attention à l’étanchéité de la plaque.
4. Ajouter le tampon TAE 1X jusqu’à recouvrir le gel d’agarose.
5. Préparer les échantillons.
Mélanger 5µL de tampon de charge pour 15µL d’échantillon d’ADN.
Préparer le marqueur de taille moléculaire si besoin :
High range : 0,5µL d’ADN + 1µL Tampon de charge du marqueur + 4,5µL d’H2O bi distillée.
6. Vortexer puis centrifuger 2 secondes.
7. Déposer délicatement les échantillons et le marqueur de masse moléculaire.
Ne pas utiliser les puits situé sur les coté du gel.
Déposer le marqueur de masse moléculaire au centre du gel pour faciliter l’analyse de la taille des échantillons.
Une plaque 8 puits a une contenance d’environ 45µL d’échantillon.
Une plaque 15 puits peut contenir 15µL d’échantillon.
7. Placer les électrodes de la cuve d’électrophorèse en sorte que l’ADN migre de l’anode vers la cathode.
8. Déclencher la migration du gel de 85 volts à 130 volts pendant 45 à 80 minutes.
Vérifier qu’il y a bien apparition de bulles due au courant électrique et que l’ADN migre dans le bon sens.
Extraction d’ADN à partir d’un gel d’agarose (Nucleospin Clontech)
Matériel
30ml de tampon NT
14ml de tampon NT3
15ml de tampon NE
50 colonnes Nucleospin Extract II
50 tubes collecteurs Nucleospin (2ml)
Scalpel stérile
Balance
Vortex
Incubateur (50°C)
Centrifugeuse (11000g)
Tubes 1.5ml stérile
Procédure
1. Exciser un fragment du gel d’agarose contenant le fragment d’ADN, avec le scalpel, en prenant soin de réduire au minimum le volume de gel.
2. Déterminer le poids de la tranche de gel et la transférer à un tube stérile.
3. Pour chaque 100 mg gel d'agarose ajouter 200μl de tampon NT.
Pour les gels contenant plus de 2% d'agarose, doubler du volume de tampon NT.
Le poids maximal par tranche de gel, par NucleoSpin® Extract II, est de 400 mg ou 200 mg pour les pourcentages élevés (> 2%), dans ce cas, 2 étapes sont nécessaires (étapes de liaison de l’ADN : 6 et 7).
4. Incuber les échantillons à 50 ° C jusqu'à ce que le gel soit dissout (5-10 min).
5. Vortexer l'échantillon brièvement tous les 2 à 3 min jusqu'à ce que le gel soit complètement dissout.
6. Placer une colonne NucleoSpin ® Extrait II dans un tube collecteur Nucleospin de 2 ml et mettre l'échantillon, puis centrifuger pendant 1 min à 11000g.
7. Jeter la solution contenue dans le tube collecteur Nucleospin et replacer la colonne NucleoSpin Extract II ® dans le même tube collecteur Nucleospin.
8. Ajouter 600μl de tampon NT3 et centrifuger pendant 1 min à 11000g.
9. Jeter la solution contenue dans le tube collecteur Nucleospin et replacer la colonne NucleoSpin Extract II ® dans le tube collecteur Nucleospin.
10. Centrifuger pendant 2 min à 11000g pour supprimer le tampon NT3.
Assurez-vous que la colonne Nucleospin ne soit pas en contact avec la solution éluée lors de la centrifugation (dans le tube collecteur Nucleospin).
L'éthanol résiduel issu du tampon NT3 inhibe les réactions ultérieures et doit être retiré à cette étape. En plus de la centrifugation, une suppression totale peut être réalisée à partir d’une incubation de la colonne NucleoSpin ® Extrait II pendant 2-5 min à 70°C avant l'élution.
11. Placer la colonne NucleoSpin ® Extract II dans un tube propre de 1,5 ml.
12. Ajouter 15-50μl de tampon d'élution NE, puis incuber à température ambiante pendant 1 min pour augmenter le rendement de l'ADN élué.
13. Centrifuger pendant 1 min à 11000g.
Le rendement des grands fragments (> 5-10 kb) peut être augmenté par l'utilisation de tampon d'élution préchauffé (70 ° C): Pour l'élution, ajouter des tampons d'élution préchauffée et incuber à température ambiante pendant 1 min avant de recueillir l'éluant par centrifugation.
Préparation d’Escherichia coli électrocompétentes
Matériel
500µl de culture d’E.Coli dans du milieu de culture LB
Milieu de culture LB
60ml de glycérol à 10%
Ethanol
Incubateur avec rotation
Bac de Glace
Tubes de 1.5ml
Tubes Falcon 50ml
Tubes pour centrifugeuse 300ml
Centrifugeuse réfrigérée à 4°C
Procédure
0. Préparer une pré-culture d’E.Coli dans 5ml de milieu de culture LB (toute la nuit à 37°C avec rotation).
Préparer la centrifugeuse à 4°C
1. Mettre la pré-culture d’E.Coli dans 450ml de milieu de culture LB et incuber à 37°C avec agitation pendant 3 heures.
2. Placer 225ml de la solution dans 2 tubes pour centrifugeuse.
3. Placer les deux tubes dans la glace pendant 15 minutes.
4. Centrifuger à 2000g pendant 10 minutes à 4°C.
7. Eliminer le surnageant et remettre délicatement en suspension avec 200ml d’eau froide stérile.
Ajouter d’abord 10 à 20ml d’eau froide stérile et resuspendre par pipetage, puis ajouter le reste des 200ml.
8. Centrifuger à 2000g pendant 10 minutes à 4°C.
9. Eliminer le surnageant et remettre délicatement en suspension avec 200ml d’eau froide stérile.
Ajouter d’abord 10 à 20ml d’eau froide stérile et resuspendre par pipetage, puis ajouter le reste des 200ml.
10. Placer les tubes dans la glace pendant 30 minutes.
11. Centrifuger à 2000g pendant 15 minutes à 4°C.
12. Eliminer le surnageant et remettre délicatement en suspension avec 25ml de glycérol à 10%.
On peut éventuellement transférer la solution dans un tube Falcon de 50ml.
13. Placer les tubes dans la glace pendant 30 minutes.
14. Centrifuger à 1500g pendant 15 minutes à 4°C.
15. Eliminer le surnageant et ajouter 500µl de glycérol à 10%.
16. Resuspendre les cellules dans un volume total de 1ml.
17. Aliquoter 50µl par tube (tubes déjà dans la glace).
18. Stocker à -80°C
Electroporation d’Escherichia coli électrocompétentes
Matériel
Milieu LB liquide
Milieu LB solide (milieu de culture LB + agarose + antibiotique adapté)
Bactéries Escherichia coli électrocompétentes congelées (40 microlitres dans un tube eppendorf)
ADN
Bac rempli de glace
Cuvettes d’électroporation (2 mm)
Electroporateur
Etuve avec agitation
Procédure
0. Placer la cuvette d’électroporation dans de la glace.
1. Mettre le tube contenant les bactéries (environ 40 microlitres) à température ambiante, afin de dégeler le milieu. Le placer ensuite dans de la glace.
2. Régler l’électroporateur avec les paramètres suivants :
Voltage 2500 V
Capacitance 25 µF
Résistance 200 Ω
Cuvette 2 mm
3. Ajouter un microlitre de la solution d’ADN au tube contenant les bactéries.
4. Placer le tube une minute dans la glace.
5. Transférer le contenu du tube dans la cuvette d’électroporation.
6. Sécher l’extérieur de la cuvette avec du papier absorbant. La mettre dans le compartiment prévu de l’électroporateur.
7. Déclencher le pulse.
8. Rajouter immédiatement 1mL de milieu de culture dans la cuvette.
Attention : un retard de 3 minutes diminue l’efficacité de la transformation de 90%.
9. Transférer le contenu de la cuvette dans un tube. Le mettre à incuber une heure à 37°C sous agitation.
Tremper les cuvettes quelques minutes dans de l’eau de javel. Les rincer abondamment avec de l’eau du robinet, puis avec de l’eau bidistillée et de l’éthanol à 70% (pas d’acétone), avant de les laisser sécher. Elles pourront ainsi être réutilisées.
10. Mettre les bactéries en culture sur un milieu LB solide.
Préparation de Mycobacterium électrocompétentes
Matériel
500μl de Culture de Mycobacterium à -80°C
Glycérol 10%
Tubes de 1.5ml
Centrifugeuse
Procédure
0. Toute la manipulation se déroule et tous les milieux sont à température ambiante.
1. Transférer les cellules de votre échantillon congelé dans des tubes de centrifugation.
2. Centrifuger les cellules à 3600rpm pendant 10 min, puis éliminer le surnageant.
3. Laver les cellules avec 1/2 volume (25ml) du glycérol 10% stérile en pipetant doucement jusqu’à ce que la masse de cellules soit dissoute.
4. Laver et culotter les cellules comme l’étape 2 et 3; avec 1/4 vol (12.5ml) de glycérol 10% stérile.
5. Laver et culotter les cellules comme l’étape 2 et 3; avec 1/8 vol (6,75ml) de glycérol 10% stérile.
6. Laver et culotter les cellules comme l’étape 2 et 3; avec 1/10 vol (5ml) de 10% glycérol stérile.
7. Laver et culotter les cellules comme l’étape 2 et 3; avec 1/25 vol (2ml) de 10% glycérol stérile.
8. Aliquoter 100μL de cellules dans des tubes de 1,5ml.
9. Laisser refroidir rapidement dans un bain de glace.
10. Puis stocker à -80°C, ou utiliser immédiatement (efficacité de l’électrotransfert augmentée).
Electroporation de Mycobacterium électrocompétentes
Matériel
Milieu Loewenstein liquide
Milieu Loewenstein solide (milieu de culture Loewenstein coagulé + antibiotique adapté)
Mycobacterium électrocompétentes congelées (40 microlitres dans un tube)
ADN
Bac rempli de glace
Cuvettes d’électroporation (2 mm)
Electroporateur
Etuve avec agitation
Procédure
0. Placer la cuvette d’électroporation dans de la glace.
1. Mettre le tube contenant les bactéries (environ 40 microlitres) à température ambiante afin de dégeler le milieu. Le placer ensuite dans de la glace.
2. Régler l’électroporateur avec les paramètres suivants :
Voltage 2500 V
Capacitance 25 µF
Résistance 1000 Ω
Cuvette 2 mm
3. Ajouter un microlitre de la solution d’ADN au tube contenant les bactéries.
4. Placer le tube une minute dans la glace.
5. Transférer le contenu du tube dans la cuvette d’électroporation.
6. Sécher l’extérieur de la cuvette avec du papier absorbant. La mettre dans le compartiment prévu de l’électroporateur.
7. Déclencher le pulse.
8. Rajouter immédiatement 1mL de milieu de culture dans la cuvette.
Attention : un retard de 3 minutes diminue l’efficacité de la transformation de 90%.
9. Transférer le contenu de la cuvette dans un tube. Le mettre à incuber une heure à 37°C sous agitation.
Tremper les cuvettes quelques minutes dans de l’eau de javel. Les rincer abondamment avec de l’eau du robinet, puis avec de l’eau bidistillée et de l’éthanol à 70% (pas d’acétone), avant de les laisser sécher. Elles pourront ainsi être réutilisées.
10. Mettre les bactéries en culture sur un milieu Loewenstein solide.
Restriction
Matériel
10µL d’ADN (1µL pour 0,5µg d'ADN)
3µL de tampon de restriction (1X du volume total)
1µL d’enzyme de restriction
3µL BSA (1X du volume total)
15µL d’eau distillée stérile
Volume Total 30µL
Tubes de 1.5ml
Procédure
1. Dans un tube eppendorf stérile.
2. Mettre Tampon 3µL de tampon.
3. Ajouter l'ADN.
4. Ajouter 3µL de BSA.
5. Puis, ajouter 1µL d’enzymes de restriction.
6. Et compléter avec l’eau distillée stérile.
VOLUME TOTAL = 30µL.
7. Mettre dans un thermocycler avec les conditions:
a. 1 - 2 H à 37°C,
b. 20 min à 80°C pour l'inactivation des enzymes,
c. Laisser à 4°C jusqu'à ouverture du thermocycler.
NOTE : si on peut laisser plus de 6h d'incubation pour la restriction, laisser plus longtemps pour avoir une restriction de meilleure qualité!
Linéarisation
Matériel
Template d’ADN
1µl de phosphatase alcaline
3µl de tampon de phosphatase alcaline
Eau Pure Volume total 30µl
Bain marie ou plaque chauffante
Tubes de 1.5ml
Procédure
0. Préparer un bain marie à 37°C.
1. Ajouter le tampon de la phosphatase alcaline concentration 1X dans le template d’ADN, soit 3µL de tampon pour 30µL de volume final.
2. Ajouter 1µL d’alkaline phosphatase.
3. Incuber 1 heure à 37°C.
4. Inactivation par la chaleur de la phosphatase alcaline 5 minutes à 65°C.
Procéder à la ligation juste après la linéarisation.
Ligation
Matériel
3µl de tampon de ligation 10X
1µl de T4 DNA Ligase
100 à 1000µg en solution d’insert d’ADN
1µl de vecteur d’ADN à 0.05g/l
Eau stérile pour un volume final de 30µl
Tubes de 1.5ml
Procédure
1. Dans un tube de 1,5ml, mettre 2µl de tampon de ligation.
2. Ajouter l’insert d’ADN ainsi que 1µl de vecteur.
3. Ajouter 1µl de T4 DNA Ligase.
4. Compléter avec de l’eau stérile pour avoir 30µl de volume final.
5. Laisser agir pendant 35 minutes à 22.5°C.
6. Dénaturer la T4 DNA Ligase à 65°C pendant 10 minutes.
PCR (Kit Promega)
Matériel
Template d’ADN
Primer 5’ à 3’
Primer 3’ à 5’
GoTaq Hot Start Green Mix (Promega)
Thermocycler
Procédure
1. Mettre le kit GoTaq Hot Start Green mix à température ambiante, vortexer puis centrifuger brièvement pour concentrer le mix au fond du tube.
2. Préparer le mix de PCR suivant :
a. Pour 25µL :
b. Pour 50µL :
c. Pour 100µL :
3. Ajouter dans l'ordre donné dans les tableaux.
Faire attention à bien insérer les volumes de primers, de template sur les bords du tube afin de voir tous les volumes déposés.
4. Vortexer le volume total
5. Centrifuger "Start - stop" (2 à 5 secondes)
Si le thermocycler n'a pas de couvercle chauffant ajouter 1 à 2 gouttes d'huile minérale stérile.
6. Insérer la préparation dans le thermocycler, puis lancer les cycles
7. Vérifier vos produits de PCR par électrophorèse.
 "
"