Team:SupBiotech-Paris/Ciblage Cellulaire
From 2009.igem.org
(→Lignée cancéreuse et gène apporté) |
Enguerrand (Talk | contribs) |
||
| Line 1: | Line 1: | ||
| - | {{Template: | + | {{Template:Supbiotechcss12.css}} |
{{Template:SupbiotechparisFr}} | {{Template:SupbiotechparisFr}} | ||
Revision as of 13:20, 19 October 2009
Contents |
Mise en application du plasmide thérapeutique sur le cancer du poumon
Contexte
Dans le cancer du poumon non à petites cellules, ou NSCLC, comme dans tous cancers, la perte de la capacité apoptotique des cellules tumorales est du à la perte fonctionnelle de divers suppresseurs de tumeur entrant dans la voie de signalisation de la cascade apoptotique.
L’application du DVS dans la lutte anti-cancer repose sur le fait de réactiver cette cascade apoptotique en apportant au sein des cellules tumorales une version wild-type des gènes codant les suppresseurs de tumeur non-fonctionnels.
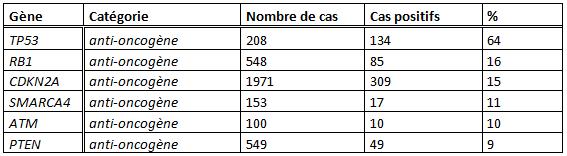
C’est le [http://www.sanger.ac.uk/genetics/CGP/cosmic/ projet COSMIC] de [http://www.sanger.ac.uk/ l’institut Sanger] qui nous a permis de déterminer quels gènes apporter au plasmide thérapeutique dans le cadre du cancer du poumon à non petites cellules. Ce projet répertorie en effet toutes les mutations détectées pour chaque type de cancers suivant leur fréquence d’apparition. Ainsi, d’après leurs données, la perte de la capacité apoptotique des cellules tumorales pour un cancer du poumon peut être du à la perte fonctionnelle des protéines issus des gènes suivant :
Ces différents gènes, jouant un rôle prépondérant dans la mise en place du processus apoptotique et étant les plus susceptibles d’avoir mutés dans le cadre d’un cancer du poumon, compose le plasmide thérapeutique.
L’objectif
L’objectif de cette étude est de vérifier si le fait d’amener une version wild-type d’un gène suppresseur de tumeur au sein d’une cellule tumorale pour qui sa version est mutée, induit ou pas le phénomène d’apoptose.
Démarche expérimentale
Lignée cancéreuse et gène apporté
Nous avons sélectionné parmi les lignées cellulaires qui étaient à notre disposition, une lignée cancéreuse dont l’origine cancéreux était du à la mutation d’un gène suppresseur de tumeur. La version wild-type du gène TP53 étant en notre possession, c’est la lignée cancéreuse prostatique p53 muté DU-145 qui retint notre attention.
Nous allons donc tester si le fait d’amener une version wild-type de la protéine p53 (p53wt) au sein de la lignée DU-145 permet le déclenchement du processus d’apoptose.
Protocole de mise en culture :
- Sortir l’ampoule de l’azote liquide
- Placer l’ampoule dans un bain-marie à 37°C pendant 5 minutes
- Dans un falcon 50 ml, mettre 9 ml de MEM 10% + 1 ml d’ampoule
- Centrifuger 5 min à 1200 rpm
- Aspirer le surnageant sans toucher aux cellules culotées (élimination du DMSO)
- Resuspendre le culot dans 1 ml de milieu
- Déposer le tout dans un nouveau T25 contenant 5 ml de milieu
- Incubation à 37°C
- Ne pas oublier de changer le milieu le lendemain pour éliminer les traces de DMSO
- Après une semaine, les cellules sont à confluence 100%
Incorporation du gène TP53
L’incorporation du plasmide contenant p53wt, pcDNA3 CMV+p53wt, au sein des cellules DU-145 s’est effectuée par électroporation.
Matériel :
- Cellules DU-145
- Plasmide pcDNA3 CMV+p53wt
- Milieu de culture électrocompétent
- Trypsine
- PBS
- Bac à glace
- Cuvette d’électrotransfert
- Centrifugeuse
- Incubateur
- Electroporateur (cliniporateur)
Protocole:
- Aspirer le milieu du T25 contant les DU-145
- Rincer au PBS
- Déposer 500 µl de trypsine et laisser agir 3 minutes à température ambiante
- Ajouter 5 ml de MEM 10% pour neutraliser la trypsine
- Suspendre les cellules
- Récupérer le milieu contenant les DU-145 dans un tube et centrifuger à 1000rpm pendant 10 minutes
- Aspirer le surnageant et resuspendre le culot dans Xµl (X= 90µl x Nombre de cuves) de milieu électrocompétent (environ 5x105 cellules par cuves)
- Suspendre votre solution d’ADN dans du milieu électrocompétent (18x10-2g/L)
- Ajouter 10µl de solution d’ADN par cuve
- Ajouter 90µl de la suspension cellulaire
- Mettre les cuves dans la glace
- Passer les cuves à l’électroporateur (cliniporateur) et enregistrer chaque résultat
- Incuber les cuves à 37°C pendant 30 minutes
- Mettre le contenu de chaque cuve dans un tube stérile, ajouter 3ml de milieu de culture MEM 10%, puis incuber à 37°C pendant le temps nécessaire (jusqu’au test à l’annexine V)
Détection de l’apoptose
La détection des cellules apoptotiques s’est effectuée par le test à l’annexine V :
En phase précoce de l’apoptose, on observe la translocation de la phosphatidyl-sérine à l’extérieur de la membrane plasmique. Celle-ci est mise en évidence par fixation spécifique de l'annexine V couplée à un fluorophore et analysée par cytométrie en flux.
Matériel :
- Iodure de propidium 1 mg/ml In vitrogen conservé au frigidaire à diluer 10 fois
- Annexine V
- Tampon annexine
Travailler le plus possible dans l’obscurité (fluorophore photolabile)
Protocole :
- Récupérer le milieu de culture (3 ml), le déposer dans un falcon 50 ml
- Rincer la culture avec 3 ml de PBS, les déposer dans le falcon
- Décoller les cellules à la trypsine, les déposer dans le falcon
- Centrifuger
- Reprendre le culot dans 0.5 ou 1 ml de PBS froid en fonction du niveau de confluence
- Prélever 10 µl pour un comptage et centrifuger
- Re-suspendre le culot dans du tampon annexine à la concentration de 1*106 cellule/ml
- Pipetter 2 aliquots de 100 µl dans 2 tubes FACS
- Ajouter dans chaque tube 5 µl d’annexine V et 1 µl de iodure de propidium
- Incuber 15 min à RT
- Arrêter la réaction en plaçant les tubes dans la glace fondante
- Ajouter 400 µl de tampon d’annexine V
- Lire au FACS le plus rapidement possible en conservant les tubes dans la glace
Déroulement de l’étude
Ne connaissant pas le temps d’expression du plasmide au sein de la lignée DU-145, nous avons réalisé un suivi cinétique de l’induction de l’apoptose en pratiquant un test à l’annexine V toutes les 6 heures pendant 48h après son électroporation. De ce fait, en couplant les taux d’apoptose de la population témoin (électroporation à vide) et de la population test (électroporation avec plasmide) avec leur taux de croissance respectifs, nous serons en mesure de déterminer l’impacte de p53wt sur l’induction de l’apoptose. La population témoin permettant d’éliminer les morts cellulaires dus à l’électroporation et au transfert de culture.
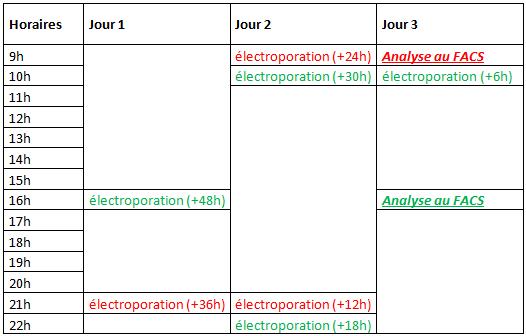
N’ayant pas eu un accès continu au cytomètre en flux, nous avons regroupé l’ensemble des 48h d’analyse en deux runs de cytométrie. Chaque créneau horaire de l’étude est représenté par une population cellulaire distincte. Ainsi nous avons réalisé 14 électroporations correspondant aux 7 créneaux horaires : +6h, +12h, +18h, +24h, +30h, +36h et +48h (deux par créneaux : population test + population témoin).
Voici le planning de répartition des électroporations:
Trois populations cellulaires ont donc été respectivement électroporées 12h, 24h et 36h avant le premier run de cytométrie (en rouge, à 9h, jour 3), quatre autres 6h, 18h, 30h et 48h avant le second run (en vert, à 16h, jour 3).
La première analyse cytométrique nous a permis d’obtenir les données pour le suivi à +12h, +24h et +36h, tandis que la seconde, nous a permis d’obtenir les données pour le suivi à +6h, 18h, +30h et +48h.
En couplant toutes ces données, on obtient un suivi sur 48h de l’induction de l’apoptose après électroporation de p53wt.
Résultats
Chaque population cellulaire représentant les différentes tranches horaires du suivi a subi un test à l’annexine V à l’instant escompté. Voici donc les résultats obtenus 6h, 12h, 24h, 30h, 36h et 48h après électroporation de p53wt :
Electroporation à vide (témoin) :
Graphe Cytométrie :
Tableau des données :
Electroporation du plasmide :
Graphe cytométrie :
Tableau des données :
File:Données électroporation.jpeg
Résultats globaux :
-
Courbes:
- Nombre de cellule = f (t)
- % apotose = f (t)
Conclusion
 "
"