Team:Paris/Addressing testing
From 2009.igem.org
(→Adressing system) |
David.bikard (Talk | contribs) (→WetLab) |
||
| Line 6: | Line 6: | ||
<center> [[team:Paris/WetLab#WetLab| Main]] - [[Team:Paris/Addressing_testing#top|Addressing]] - [[Team:Paris/Production_testing#top| Production]] - [[Team:Paris/Transduction_testing#top | reception]] - </center> | <center> [[team:Paris/WetLab#WetLab| Main]] - [[Team:Paris/Addressing_testing#top|Addressing]] - [[Team:Paris/Production_testing#top| Production]] - [[Team:Paris/Transduction_testing#top | reception]] - </center> | ||
<center> '''Addressing'''</center> | <center> '''Addressing'''</center> | ||
| + | |||
| + | |||
| + | <font color=yellow>Ne vous prenez pas trop la tête avec la description des constructions. Il faut surtout faire des liens vers nos parts à la registry</font> | ||
<html> | <html> | ||
</div> | </div> | ||
| Line 12: | Line 15: | ||
<div id="paris_content"> | <div id="paris_content"> | ||
</html> | </html> | ||
| - | |||
== Adressing system == | == Adressing system == | ||
Revision as of 15:34, 18 October 2009
iGEM > Paris > WetLab > Addressing
Contents |
WetLab
Ne vous prenez pas trop la tête avec la description des constructions. Il faut surtout faire des liens vers nos parts à la registry
Adressing system
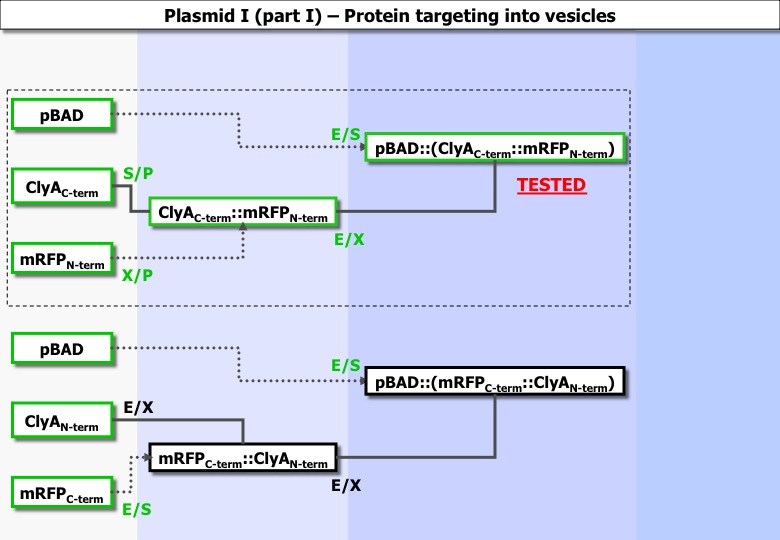
global constructions :
Time required : A lOT !!!!!
Experiments ran :
| column 1 | column 2 | column 3 | column 4 |
| PCR :
ClyA Cterm matrix : ? Oligo : TM : ClyA Nterm matrix : ? Oligo : TM : mRFP Cterm matrix : ? Oligo : TM : mRFP Nterm plasmid : ? Oligo : TM : | PCR :
- | PCR :
- | PCR :
- |
| Verification on gel :
ok | Verification on gel :
- | Verification on gel :
- | Verification on gel :
- |
| Purification on gel :
ok | Purification on gel :
- | Purification on gel :
- | Purification on gel :
- |
| digestion:
ClyA Cterm S/P RFP Nterm X/P PBAD vector : ? E/S
| digestion:
ClyA Cter-RFP Nter vector PSB1A3 E/X | digestion:
- | digestion:
- |
| verification digestion:
ok | verification digestion:
ok | verification digestion:
- | verification digestion:
-
|
| ligation:
ClyA Cter-RFP Nter vector PSB1A3 | ligation:
PBAD E/S ClyA Cter-RFP Nter PSB1A3 E/X (x2) PBAD S/P ClyA Cter-RFP Nter PSB? X/P (x2) | ligation:
- | ligation:
- |
| PCR colo :
ok | PCR colo :
ok | PCR colo :
- | PCR colo :
-
|
| miniprep:
clone : | miniprep:
clone : | miniprep:
- | miniprep:
-
|
| sequencing :
ok | sequencing :
ok | sequencing :
- | sequencing :
- |
| stock glycerol:
S? S? | stock glycerol
S? | stock glycerol
- | stock glycerol
-
|
Functional Testing:
PBAD ClyA RFP was transformed into Top10 bacteria in order to localize the fluorescence, we are supposed to have a superior fluorescence in the membrane.
PBAD ClyA RFP on PSB3T5 was transformed into Delta Tol bacteria , in this case we are supposed to see fluorescent vesicles went the medium contains 1 % arabinose , and to have no fluorescent on 1% glucose.
Export system
We finally thought that it won't be neccesary to overexpress the Tat system, nevertheless we have run a few experiments before starting to focus on others parts of the project.
Time required : A week.
Experiments ran :
| column 1 | column 2 | column 3 | column 4
|
| PCR :
TatABCE matrix : | PCR : | PCR : | PCR : |
| Verification on gel :
ok | Verification on gel :
- | Verification on gel :
- | Verification on gel :
- |
| Purification on gel :
ok | Purification on gel :
- | Purification on gel :
- | Purification on gel :
-
|
| digestion:
TatABCE | digestion: | digestion: | digestion: |
| verification digestion:
ok | verification digestion:
- | verification digestion:
- | verification digestion:
-
|
| ligation:
TatABCE | ligation: | ligation: | ligation: |
| PCR colo :
ok | PCR colo :
- | PCR colo :
- | PCR colo :
- |
| miniprep:
STOPPED | miniprep:
- | miniprep:
- | miniprep:
- |
| sequencing :
- | sequencing :
- | sequencing :
- | sequencing :
- |
 "
"